Nat Commun:可解释AI模型regX揭秘细胞状态转移的驱动因子和调控机制
时间:2025-02-07 12:12:24 热度:37.1℃ 作者:网络
在生物学和医学研究中,理解细胞状态转移的调控机制一直是一个复杂而关键的问题。识别细胞状态转移过程中的驱动调控因子对于理解细胞命运决定的分子机制、实现细胞状态控制具有重要意义。
近日,清华大学张学工团队在Nature Communications发表了题为“A mechanism-informed deep neural network enables prioritization of regulators that drive cell state transitions”的文章。研究团队基于上游调控因子到靶基因、功能通路再到细胞状态的跨层次调控过程,设计了一种可解释的神经网络模型regX。通过虚拟扰动(in-silico perturbation)实验,模型能对可能驱动细胞状态转移的关键转录因子(TF)和候选顺式调控元件(cCRE)进行排序,并为其提供机制解释。论文第一作者是清华大学自动化系博士生席熙(已毕业)。

文章发表于Nature Communications
regX用两级异构的神经网络实现从cCRE和TF到细胞状态的预测,利用scRNA-seq和scATAC-seq数据进行训练,基因转录调控、蛋白质相互作用或功能通路作为调控细胞状态的重要中间过程嵌入网络隐层。这种生物机理引导的设计,使regX不但能识别潜在驱动调控因子,还能解释这些因子如何通过调控下游靶基因和功能通路影响细胞状态转移。
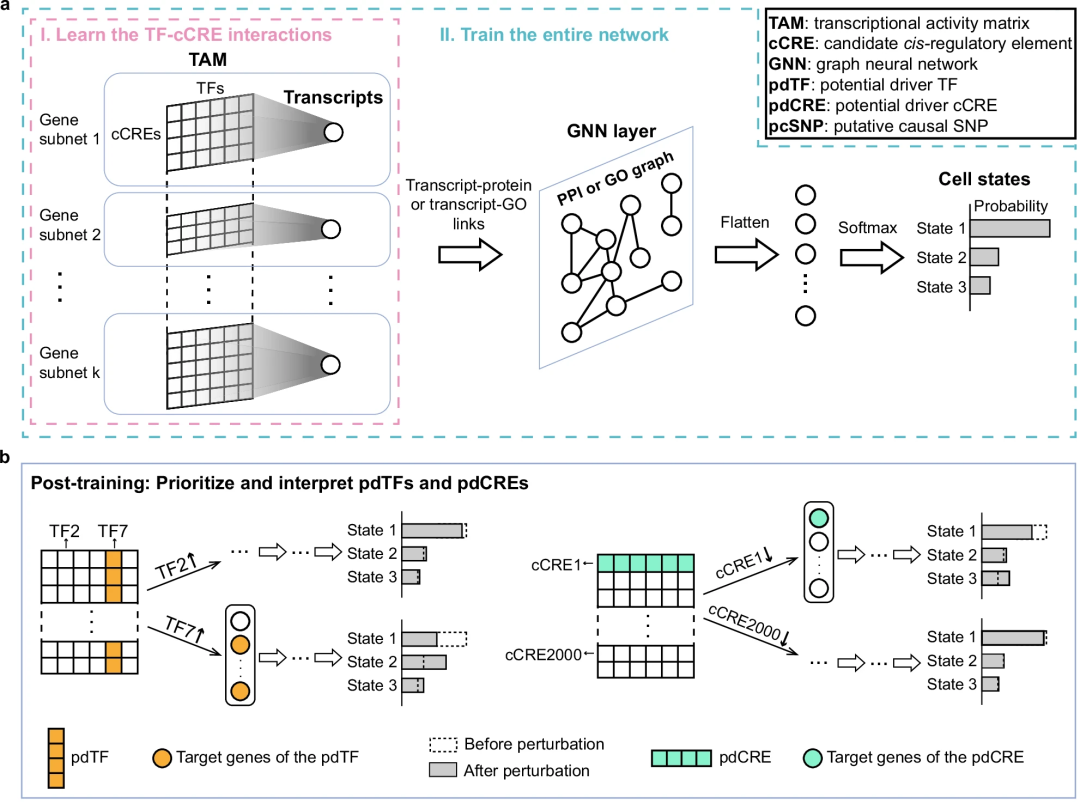
regX的模型结构、训练与推理
研究团队将regX应用于两个公开的单细胞多组学数据集:二型糖尿病(T2D)和毛囊发育。在T2D数据集上,regX识别出胰岛β细胞中与T2D进展相关的关键TF和cCRE,并揭示了与T2D相关的潜在治疗靶点、药物重定位机制和两类潜在因果变异。在毛囊发育数据集上,regX展示了其在量化调控因子对细胞命运决定中的优势,发现同一转录因子的不同上调水平可能导致不同的细胞命运决定。
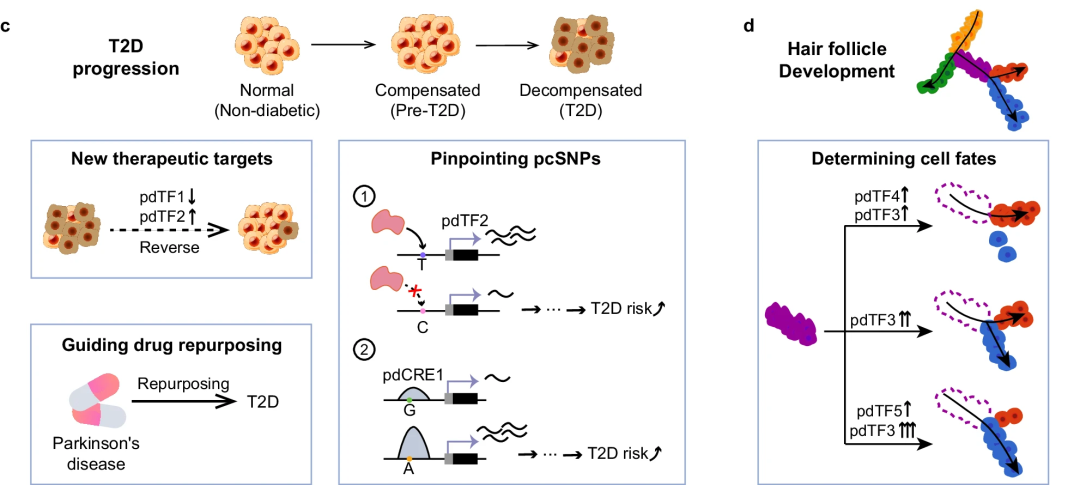
regX的应用案例
regX为研究疾病或发育过程中的细胞状态转移及其调控提供了有力工具。用户可根据需求,利用公开或自测的单细胞多组学数据集进行模型训练和推理,模拟TF上下调以及cCRE区域开放或关闭对细胞状态的影响,对潜在驱动TF和cCRE进行排序并揭示其调控机制。
regX的详细使用教程和代码见网页https://github.com/xixi-cathy/regX/。
论文原文链接:https://doi.org/10.1038/s41467-025-56475-9


