【论著】| 胰腺癌吉西他滨化疗耐药生物标志物—INPP4B的探索性研究
时间:2025-01-24 12:08:49 热度:37.1℃ 作者:网络
[摘要] 背景与目的:胰腺癌是具有高侵袭性的消化系统实体肿瘤,因病程初期缺乏特异性临床表现,约80%的患者无法行根治性切除术。吉西他滨作为胰腺癌化疗的一线用药,由于化疗耐药性,其使用并未显著改善患者预后。迄今,胰腺癌对吉西他滨治疗发生耐药的具体机制仍不清楚。本研究借助胰腺癌吉西他滨耐药细胞系及胰腺癌病理组织标本,结合在线数据库,探索胰腺癌吉西他滨化疗耐药的潜在生物标志物。并通过收集与分析胰腺癌患者的随访资料,明确相关靶点对胰腺癌患者预后的影响。方法:本研究采用吉西他滨浓度递增法逐步诱导构建胰腺癌耐药细胞株;对耐药细胞行二代高通量RNA-seq测序,并联合在线网络数据库高通量基因表达数据库(Gene Expression Omnibus,GEO)的4个胰腺癌吉西他滨耐药相关GEO数据集(GSE106336、GSE110580、GSE35141和GSE140077),用生物信息学分析筛选共同差异表达基因;采用实时荧光定量聚合酶链反应(real-time fluorescence quantitative polymerase chain reaction,RTFQ-PCR)、蛋白质印迹法(Western blot)及免疫组织化学验证目标分子的表达水平;最后选取自2018年6月—2021年6月就诊于西安交通大学第一附属医院并经手术后病理学检查确诊为胰腺腺癌的部分患者,从西安交通大学第一附属医院生物样本信息资源中心获取行胰腺癌根治性切除术的部分患者手术标本共70例(其中30例对吉西他滨治疗耐药,16例未耐药,同时取24例癌旁胰腺组织作为对照;伦理批件号:2021伦审科字第(457)号,No.XJTU1AF2021LSK-457),并收集其临床和预后相关信息,用log-rank检验评价目标分子的表达与患者预后的关联性。结果:检测胰腺癌吉西他滨耐药细胞株(MiaPaCa-2 gemcitabine resistant,Mia GR)及同种野生型细胞系(MiaPaCa-2 wild type,Mia WT)针对吉西他滨杀伤的半数抑制浓度(half maximal inhibitory concentration,IC50),结果表明Mia GR组的IC50显著高于Mia WT组(258.10 μmol/L vs 0.18 μmol/L),耐药指数(resistence index,RI)高达1 443.9。对耐药株与野生细胞系的二代转录组测序结果分析得到3 985个差异表达基因,再与4个胰腺癌吉西他滨耐药的在线GEO数据集(GSE106336、GSE110580、GSE35141和GSE140077)中的数据作横向比较,发现有25个共同差异表达基因。结合文献回顾和对基因的功能注释和通路富集分析,进一步筛选发现INPP4B在其中发挥关键作用。接下来通过细胞学的RTFQ-PCR及Western blot检测,证实INPP4B在耐药细胞中的mRNA表达和蛋白水平均显著高于野生型(P<0.05)。最后对30例耐药胰腺癌组织、16例未耐药胰腺癌组织以及24例癌旁胰腺组织进行免疫组织化学分析。结果显示,与未耐药组相比,耐药组胰腺癌患者的INPP4B表达水平明显更高,而正常组织中INPP4B的表达水平明显低于这两种类型的癌组织。将胰腺癌组织中的INPP4B表达水平与患者生存信息相对应,绘制Kaplan-Meier曲线,结果表明,INPP4B低表达组患者的无进展生存期(progression-free survival,PFS)显著优于INPP4B高表达组(HR=2.874,95% CI:1.262~6.544,P=0.013)。同时,低INPP4B表达组患者的总生存期(overall survival,OS)相比INPP4B高表达组也有明显优势,虽然其间差异无统计学意义(HR=1.484,95% CI:0.518~4.250,P=0.465)。结论:INPP4B可能是一种潜在的胰腺癌吉西他滨化疗耐药生物标志物,且与耐药胰腺癌患者的不良预后相关。针对此靶点设计相应的检测及治疗手段,将有望早期识别对吉西他滨治疗潜在耐药的胰腺癌患者,极大地改善其预后。
[关键词] 胰腺癌;吉西他滨;化疗耐药;INPP4B;预后
[Abstract] Background and purpose: Pancreatic cancer is a highly aggressive solid tumor of the digestive system, with radical rep being unfeasible in approximately 80% of patients due to the absence of specific clinical manifestations in the early stages. The use of gemcitabine as a first-line chemotherapeutic agent has not significantly improved patient prognosis, primarily due to the development of chemoresistance. The precise mechanisms underlying gemcitabine resistance in pancreatic cancer remain unclear. This study aimed to explore potential biomarkers associated with gemcitabine chemoresistance in pancreatic cancer by utilizing gemcitabine-resistant cell lines and pathological pancreatic cancer tissues, in conjunction with data from online databases. Additionally, we analyzed follow-up data from pancreatic cancer patients to assess the impact of relevant targets on patient prognosis. Methods: In this study, gemcitabine-resistant cell lines were developed through stepwise induction using a gemcitabine concentration gradient. Second-generation high-throughput RNA-seq sequencing was conducted on these resistant cells, and bioinformatics analysis was employed to identify four pancreatic cancer genes from the Gene Expression Omnibus (GEO) datasets (GSE106336, GSE110580, GSE35141, and GSE140077). Co-expressed genes were screened using real-time fluorescence quantitative polymerase chain reaction (RTFQ-PCR), Western blot and immunohistochemistry to verify the expression levels of target molecules. Surgical specimens from 70 patients diagnosed with pancreatic adenocarcinoma at the First Affiliated Hospital of Xi'an Jiao Tong University between June 2018 and June 2021 were analyzed. These included 30 specimens resistant to gemcitabine, 16 non-resistant specimens, and 24 normal pancreatic tissues as controls. Ethical approval was obtained (Ethical approval: 2021 LunxuanKeZi No. 457, No. XJTU1AF2021LSK-457). Clinical and prognostic information was collected, and the log-rank test was used to evaluate the relationship between target molecule expression and patient prognosis. Results: The half maximal inhibitory concentration (IC50) for gemcitabine was significantly higher in the gemcitabine-resistant cell strain (Mia GR) than in the wild-type cell line (Mia WT) (258.10 μmol/L vs 0.18 μmol/L), with a resistance index (RI) of 1 443.9. Transcriptome sequencing identified 3 985 differentially expressed genes, of which 25 were shared with the GEO datasets. Further analysis highlighted INPP4B as a key gene. RTFQ-PCR and Western blot confirmed that INPP4B mRNA and protein levels were significantly elevated in drug resistant cells compared to wild-type cells (P<0.05). Immunohistochemical analysis revealed that INPP4B expression was significantly higher in drug resistant pancreatic cancer tissues compared to non-drug resistant tissues, and lower in normal tissues than in both cancerous tissue types. Kaplan-Meier curves demonstrated that patients with low INPP4B expression had significantly better progression-free survival (PFS) than those with high expression (HR=2.874, 95% CI: 1.262-6.544, P=0.013). Although patients with low INPP4B expression also showed better overall survival (OS), the difference was not statistically significant (HR=1.484, 95% CI: 0.518-4.250, P=0.465). Conclusion: INPP4B may serve as a potential biomarker for gemcitabine chemoresistance in pancreatic cancer and is associated with poor prognosis in drug resistant patients. Developing targeted assays and treatments for INPP4B could facilitate early identification of patients likely to exhibit resistance to gemcitabine therapy, thereby improving their prognosis.
[Keywords] Pancreatic cancer; Gemcitabine; Chemoresistance; INPP4B; Prognosis
胰腺癌是目前预后极差的恶性肿瘤之一,其死亡率位居所有癌症死因中的第四位[1],预计到2030年将成为全球癌症死因的第二位[2]。尽管胰腺癌治疗手段的进步使得各期患者的5年生存率略有提高,但仍然不足12%[3]。化疗是胰腺癌治疗的重要组成部分,吉西他滨作为各期胰腺癌的标准疗法用药[4],可显著改善患者的预后,然而化疗耐药问题严重制约了其疗效。因此,迫切需要寻找胰腺癌吉西他滨化疗耐药中的特定生物标志物,以改善胰腺癌患者化疗现状。
INPP4B基因编码Ⅱ型肌醇聚磷酸盐4- 磷酸酶,通过调控磷脂酰肌醇3- 激酶(phosphoinositide3-kinase,PI3K)/蛋白激酶B(protein kinase,AKT)信号转导通路,在多种恶性肿瘤中显示出潜在的靶向治疗作用[5-6]。有研究[7-8]表明,INPP4B表达的增加与胰腺癌患者的不良临床结局相关。在胰腺癌组织中其上调表达,并通过激活AKT而在胰腺癌中发挥促癌作用。但也有研究[9]表明,INPP4B可以通过抑制AKT活性来抑制胰腺癌的上皮-间质转化和侵袭能力。目前涉及INPP4B与胰腺癌吉西他滨化疗耐药的关联性以及对于其在吉西他滨化疗耐药胰腺癌群体中的表达水平的研究鲜见报道。
因此,本研究使用吉西他滨耐药胰腺癌细胞株,通过生物信息学分析逐步筛选出可能的耐药生物标志物—INPP4B,并通过实时荧光定量聚合酶链反应(real-time fluorescence quantitative polymerase chain reaction,RTFQ-PCR)、蛋白质印迹法(Western blot)检测和免疫组织化学等方法进行验证。最后,利用患者的随访数据来探究INPP4B与吉西他滨化疗耐药胰腺癌患者预后的相关性。总之,本研究旨在探索胰腺癌吉西他滨化疗耐药的潜在生物标志物,为临床工作者在早期识别可能对吉西他滨耐药的胰腺癌患者提供参考。
1 材料和方法
1.1 实验材料及数据
1.1.1 细胞及标本收集
本实验所用人源性胰腺癌细胞系Mia PaCa-2来源于中国科学院典型培养物保藏委员会细胞库。标本获取自西安交通大学第一附属医院生物样本信息资源中心。
1.1.2 主要试剂
DMEM培养基购自美国Gibco公司,胎牛血清购自浙江天杭生物科技有限公司),吉西他滨购自美国MCE公司,以二甲基亚砜(dimethyl sulfoxide,DMSO)溶解;CCK-8试剂盒购自美国Bio-Rad公司;活性氧检测试剂盒购自上海碧云天生物技术有限公司;INPP4B二抗购自英国Abcam公司;RNA提取试剂盒购自上海飞捷生物技术有限公司;反转录合成试剂盒购自日本Takara公司。
1.1.3 临床数据
本研究临床数据来自2018年6月—2021年6月就诊于西安交通大学第一附属医院并经术后病理学检查确诊为胰腺腺癌的部分患者,胰腺癌患者的预后信息来源于西安交通大学第一附属医院肝胆外科随访办公室,所有患者均已签署随访知情同意书。随访日期截至2023年12月,所有患者均完成至少30个月的随访。具体纳入标准为:① 经病理学检查确诊为胰腺腺癌;② 确诊后于我院行进一步系统治疗;③ 所行化疗方案中含吉西他滨;④ 对化疗方案具备耐受能力且愿意配合随访。该研究已经西安交通大学第一附属医院伦理委员会审批通过(伦理编号:No.XJTU1AF2021LSK-457)。
1.2 实验方法
1.2.1 细胞培养及耐药细胞株构建
Mia PaCa-2细胞在含有10%胎牛血清的DMEM培养基中置于37 ℃、CO2体积分数为5%的环境中培养。每隔2~3天更换培养基,并在细胞生长达到90%时按1∶2或1∶3的比例进行传代,并取对数生长期的细胞进行后续实验。耐药细胞株(MiaPaCa-2 gemcitabine resistant,Mia GR)是通过逐步增加吉西他滨干预浓度来诱导野生细胞系(MiaPaCa-2 wild type,Mia WT)获得的,具体步骤如下:Mia WT细胞培养基中加入初始吉西他滨至浓度0.01 μmol/L,培养48 h后除去死亡细胞,并更换新的培养基继续培养。然后,按照2倍递增的方式逐渐增加药物浓度,直到达到100 μmol/L的浓度。经过10个月的操作,获得了对吉西他滨稳定耐受的细胞株Mia GR,并在后续培养和传代过程中定期向培养基中加入 5 μmol/L吉西他滨以维持细胞的耐药性。
1.2.2 CCK-8试验
分别制备处于对数生长期的Mia WT和Mia GR细胞的悬液,经计数后加入96孔板中,每孔约2 000个细胞,每孔加入100 μL培养基,每组设置3个复孔。在干预后的48 h,每孔中加入10 μL CCK-8试剂,在37 ℃条件下温育1 h后,使用酶标仪检测其在450 nm波长处的吸光度(D)值。
1.2.3 活性氧检测试验
将Mia WT及Mia GR细胞培养至6孔板中,在相同浓度的吉西他滨浓度下处理48 h后去除培养基,并用PBS洗涤细胞3次。使用活性氧检测试剂盒,并按照试剂说明书检测细胞内活性氧的含量。使用ZEISS荧光显微镜拍照记录活性氧的生成情况。
1.2.4 生物信息学分析
对Mia WT及Mia GR细胞系分别设置3个生物重复,使用Illumina TruSeq RNA样品制备试剂盒(美国CA公司)构建样品文库,并利用Illumina HiSeq2500平台进行PE150测序。使用DEseq2 R软件包分析差异表达基因,以log2 Fold change的绝对值≥1和q值<0.05作为判断差异基因表达显著性的标准。接着对差异表达基因进行层次聚类分析,并绘制火山图及热图。从在线数据库高通量基因表达数据库(Gene Expression Omnibus,GEO)(http://www.ncbi.nlm.nih. gov/geo/)下载并提取了4个GEO数据集(GSE106336[10]、GSE110580[11]、 GSE35141[12]和GSE140077[13]),用于比较不同胰腺癌耐药细胞系间的差异表达基因。使用R软件包VennDiagram(version 1.6.20)中的calculate. overlap函数,https://www.r-project.org/)获取不同组别差异基因之间的Overlap信息,并绘制韦恩图,以获得共同的差异表达基因。使用DAVID数据库(https://david.ncifcrf.gov/)进行基因功能注释(Gene Ontology,GO)和京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)通路富集分析。利用在线平台GEPIA2(http://gepia.cancer-pku.cn/index. html)分析癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库中INPP4B的表达情况。1.2.5 免疫组织化学分析
将获取的胰腺组织用福尔马林固定并包埋成蜡块,根据临床信息分布为微阵列组织芯片并切片,包括耐药和未耐药的胰腺癌组织以及癌旁胰腺组织。其中耐药标本取自吉西他滨治疗耐药组胰腺癌患者,而未耐药标本相应地取自未耐药组患者。参考实体肿瘤疗效评价准则(The Response Evaluation Criteria in Solid Tumors,RECIST)1.1版本[14]。对吉西他滨耐药定义为:开始使用吉西他滨治疗后的6个月内评估出现疾病进展(progressive disease,PD);而吉西他滨治疗开始后的6个月内表现为疾病稳定(stable disease,SD)、完全缓解(complete response,CR)或部分缓解(partial response, PR)被视作未耐药,即治疗有效[15]。考虑到吉西他滨的一个治疗周期为1个月,而6个月(即6个周期)的临界值取自各分期胰腺癌患者平均总生存期(overall survival,OS)的中位值,约12.6个月[16]。对切片进行INPP4B的免疫组织化学染色:先与抗INPP4B一抗温育,阴性对照无一抗培养。使用DAB染色,再采用苏木精复染。在切片扫描后,对每个截面在20倍放大时随机捕获3个视野进行评估。使用Image J软件检测INPP4B的染色,根据阳性细胞百分比和染色强度(转换为平均光密度)进行免疫组织化学评分。
1.2.6 RTFQ-PCR试验
从肿瘤细胞中提取总RNA,加入20 μL DEPC进行溶解,测定260 nm处D值进行定量后,反转录合成cDNA。以GAPDH为内参,采用2-ΔΔCT法分析INPP4B的基因表达水平。引物序列顺义链为:GTCTGACAAGATACTCCAGCAC,反义链为GGGAATGTGACACCAGTCAAAA。实验重复3次,取平均值。
1.2.7 Western blot检测
将细胞裂解后提取各组细胞的总蛋白,采用二辛可宁酸(bicinchoninic acid,BCA)进行蛋白定量。调齐各组蛋白浓度后加入上样缓冲液(loading buffer),并在100 ℃下加热处理10 min使蛋白变性。随后制胶、上样、电泳、转膜,并在专用封闭液封闭后加入一抗(1∶1 000稀释),在4 ℃下温育过夜。之后用吐温-20三乙醇胺缓冲盐溶液(tris-buffered saline Tween, TBST)洗膜3次,每次10 min,然后加入二抗(1∶50 000稀释),在室温下温育2 h后再次洗膜。最后加入显影剂进行曝光显影,检测INPP4B的蛋白表达。
1.3 统计学处理
定量资料符合正态分布者采用x±s表示,否则以中位数(范围)表示。对数据进行正态性检验和方差齐性分析。如果数据符合正态性和方差齐性,则采用Student-t检验,如不符合,则采用非参数检验。使用log-rank检验的Kaplan-Meier曲线分析评价OS和PFS。采用双侧检验,P<0.05为差异有统计学意义。采用SPSS 20.0软件进行统计分析,并使用GraphPad Prism 8.0软件进行图表绘制。
2 结 果
2.1 吉西他滨耐药胰腺癌细胞株的鉴定
Mia GR细胞株相比于Mia WT细胞系表现出对吉西他滨极高的耐药性。按照吉西他滨浓度梯度设置共9组,每组浓度是前一组浓度的1/2。Mia GR的浓度范围为0~200 μmol/L,而Mia WT的浓度范围为0~3 μmol/L。使用CCK-8法检测两组细胞在不同吉西他滨浓度梯度下干预48 h后的D值,并计算半数抑制浓度(half maximal inhibitory concentration,IC50)。结果显示,Mia GR组的半数致死量(median lethal dose,IC50)显著高于Mia WT组(258.1 μmol/L vs 0.18 μmol/L)。经计算,Mia GR细胞株的耐药指数(resistance index,RI)为1 443.9,表明其耐药性良好(图1)。
胰腺癌细胞在化疗药物作用下会产生活性氧,因此活性氧的产生可视作细胞对抗杀伤效应的反应指标。本研究检测了相同浓度的吉西他滨(1 μmol/L)干预后,Mia GR和Mia WT细胞内活性氧的产生情况(图1C)。细胞荧光成像结果显示,Mia WT细胞系经过吉西他滨处理后,活性氧水平明显升高,而Mia GR细胞株则活性氧水平变化不明显。从另一个角度可以看出,所构建的耐药细胞株对吉西他滨具有较强的抵抗性。

图1 吉西他滨耐药胰腺癌细胞株的鉴定
Fig. 1 Identification of gemcitabine resistant pancreatic cancer cell strain
A: IC50 quantitative efficacy curves of Mia GR; B: IC50 quantitative efficacy curves of Mia WT against gemcitabine; C: Levels of reactive oxygen species production in response to gemcitabine by Mia WT and Mia GR cell lines.
2.2 胰腺癌吉西他滨耐药细胞系的测序及结果分析
在本研究中,对两组细胞进行了高通量RNA-seq测序,结果显示,Mia GR和Mia WT之间共筛选出了3 985个差异表达基因,其中有1 559个基因上调,2 426个基因下调(图2A)。然后,使用差异倍数和差异显著性两个指标将特定的差异基因定位到坐标系中的不同位置,绘制差异表达基因的火山图(图2B),以直观地显示差异表达基因间的差异化。此外,还使用聚类分析中的K-means算法将差异表达基因划分为多个具有相似表达模式的集合,并绘制了所有组别差异表达基因的热图(图2C)。该热图的颜色差异代表了基因在不同分组间表达量(FPKM)的差异,较好地反映了样本的整体聚类结果。
为了进一步缩窄研究目标基因的筛选范围,将测序结果与采用其他胰腺癌细胞系的同类型研究在外部数据库中进行比对。与4个GEO数据集(GSE106336、GSE110580、GSE35141和GSE140077)中采用的横向比较结果相结合,发现其中有25个共同差异表达基因(图2D)。进一步对这些基因进行了功能(图2E)和通路富集分析(图2F)。结果显示,基因的表达变化在磷脂酰肌醇合成、磷酸肌醇代谢和去磷酸化等功能和通路中富集明显。结合相关的文献回 顾[17-18],INPP4B不仅可通过将PI(3,4)P2转化为PI(3)P并抑制PI3K/AKT信号转导,还可降低PI3K/AKT/SGK3信号转导通路中一些必需蛋白的表达,包括p-AKT、p-SGK3、p-mTOR、磷酸化p70S6K和PDK1等,因此进一步发掘得到上述富集的功能和通路中,均有INPP4B分子发挥着关键作用。
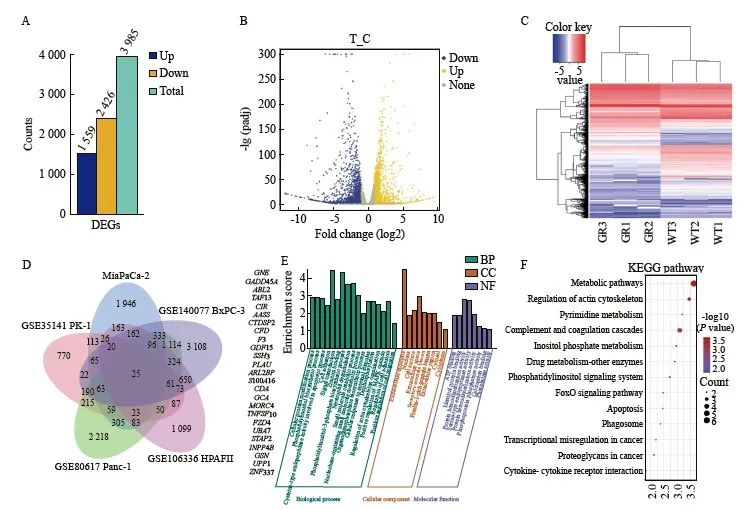
图2 胰腺癌吉西他滨耐药细胞株的测序及结果分析
Fig. 2 Sequencing of gemcitabine resistant cell strain in pancreatic cancer and analysis of the results
A: Differentially expressed genes of Mia GR and Mia WT; B: Volcano plots; C: Heat maps of differentially expressed genes; D: Common differentially expressed genes in different cell lines in the online database of isoform studies; E: Gene ontology terms of common differentially expressed genes; F: KEGG pathway enrichment analysis.
2.3 INPP4B在胰腺癌细胞及组织中的表达水平鉴定
RNA-seq测序结果显示,Mia GR细胞株中INPP4B的mRNA表达水平显著高于Mia WT细胞系(图3A)。随后进行的RTFQ-PCR验证了这一点(P<0.001,图3B)。采用Western blot检测Mia WT和Mia GR细胞中INPP4B蛋白的表达水平,结果显示,Mia GR细胞中的INPP4B蛋白表达水平同样显著高于Mia WT细胞(P<0.01,图3C)。
使用在线数据库TCGA的数据,本研究获取了179例胰腺癌患者和171例对照组的INPP4B表达情况。结果显示,胰腺癌患者中的INPP4B表达水平明显高于对照组(P<0.05,图3D),并且INPP4B的表达水平在不同分期的胰腺癌患者中存在差异(图3E)。为了进一步验证INPP4B在吉西他滨耐药胰腺癌组织中的表达情况,选取了来自本院的30例耐药组患者的胰腺癌组织、16例未耐药组患者的胰腺癌组织以及24例癌旁组织进行免疫组织化学分析。结果显示,与未耐药组相比,耐药组胰腺患者的INPP4B表达水平明显更高,而癌旁组织中INPP4B的表达水平明显低于这两种类型的癌组织(图3F),与此前得到的结果一致。

图3 INPP4B在胰腺癌细胞及组织中的表达水平鉴定
Fig. 3 Identification of INPP4B expression levels in pancreatic cancer cells and tissues
A: The mRNA expression level of INPP4B in each group of samples from sequencing results; B: RTFQ-PCR to verify the mRNA level of INPP4B in Mia GR and Mia WT; C: Western blot to detect the expression level of INPP4B in Mia WT and Mia GR and statistics; D: Difference in expression levels of INPP4B between tumor patients and control group; E: Distribution of expression levels of INPP4B in patients with different stages of pancreatic cancer in TCGA database; F: Immunohistochemical analysis of INPP4B expression levels in drug resistant pancreatic cancer tissues, non-drug resistant pancreatic cancer tissues, and para-cancerous tissues. *: P<0.05, compared with each other; **: P<0.01, compared with each other; ***: P<0.001, compared with each other.
2.4 INPP4B表达水平与胰腺癌患者预后的关系
使用获取的胰腺癌患者组织以及部分患者的生存信息(表1),本研究共计统计了46例胰腺癌患者组织中INPP4B的免疫组织化学评分,并将所得到的INPP4B表达水平与其生存信息相对应,然后绘制出相应的Kaplan-Meier曲线。
结果显示,INPP4B低表达组患者的无进展生存期(progression-free survival,PFS)显著优于高INPP4B表达组(HR=2.874,95% CI:1.262~6.544,P=0.013,图4A)。同时, INPP4B低表达组患者的OS较INPP4B高表达组也有明显优势,但差异并无统计学意义(HR=1.484,95% CI:0.518~4.250,P=0.465,图4B)。
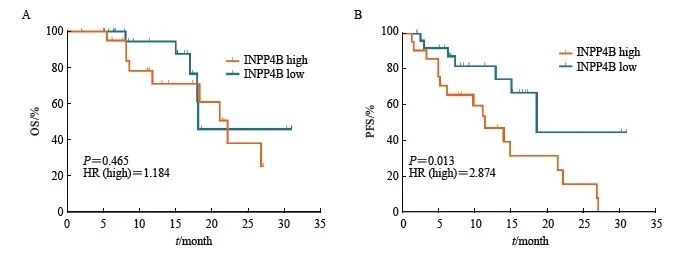
图4 INPP4B表达水平与胰腺癌患者预后的关联
Fig. 4 Association between INPP4B expression level and prognosis of patients with pancreatic cancer
A: Kaplan-Meier curve of INPP4B expression level correlated with patients’ OS; B: Kaplan-Meier curve of INPP4B expression level correlated with patients’ PFS.
3 讨 论
胰腺癌作为一类高度恶性、预后极差的疾病,在全球范围内居癌症死因的高位。目前对这种实体肿瘤唯一可能治愈的选择是根治性手术切除,但由于肿瘤高侵袭性的生物学特性和缺乏早期疾病特异性体征和症状,只有不足20%的患者在诊断时存在手术切除的可能[19]。即便接受手术,大多数患者在术后不久会面临复发和转移[20],预后仍然很差。到目前为止,化疗是有效控制胰腺癌的重要选择。吉西他滨作为胰腺癌治疗的标准用药被用于多种胰腺癌化疗方案,但往往获益有限,这在很大程度上是由于在吉西他滨治疗期间胰腺肿瘤细胞出现的耐药性所致。
关于吉西他滨耐药的原因可以被概括为肿瘤细胞的内在原因和肿瘤微环境两方面。肿瘤细胞内在原因的探索需要立足于其基因表达,不仅因为胰腺癌具有特征性的基因突变,包括常见的KRAS、CDKN2A、TP53和SMAD4[21]等,而且10%~15%的胰腺癌患者在SWI/SNF染色质重塑基因中获得与大规模结构基因组畸变相关的变 异[22],可能与其化疗耐药性有关。此外,具有免疫抑制性的肿瘤微环境可以帮助胰腺癌细胞逃避宿主的免疫监视[23],这种以癌症相关成纤维细胞、基质金属蛋白酶和独特结缔组织增生为特征的细胞外基质,被认为是对大多数单药治疗方法产生耐药性的原因[24]。目前有许多正在进行的临床试验旨在提高免疫系统对胰腺癌的敏感性,以克服肿瘤微环境的免疫抑制特性及其共有的化疗耐药性。
基因检测在胰腺癌治疗中的指导作用正在不断被强化。有研究[25]发现合并BRCA1/2突变的晚期胰腺癌患者对铂类药物敏感且能够从奥拉帕利的维持治疗中获益,基于一项前瞻性试验[26]已经批准在选择性BRCA1/2致病性胚系变异患者中使用靶向治疗药物奥拉帕利。我们的研究同样借助对Mia GR和Mia WT细胞系的测序结果,分析发现两者间大部分差异表达基因被归类为与磷酸化相关的功能和通路,如磷脂酰肌醇合成、磷酸肌醇代谢以及去磷酸化等。同时,既往研究[27]表明,吉西他滨经人体摄取后需要在细胞内磷酸化才能发挥抗肿瘤活性,且多种磷酸化反应的限速酶被证实是胰腺癌对吉西他滨耐药现象的关键分子,因此我们将研究聚焦于该类功能或通路,并进一步发掘出了其中的关键基因—INPP4B。
INPP4B基因编码一种参与磷脂酰肌醇信号转导通路的磷酸酶,其通过水解PI(3,4)P2来抑制PI3K/AKT信号转导通路的作用已得到证实[6]。PI3K/AKT信号转导通路的过度激活已被证实是肿瘤发生和发展的驱动因子[3],因此可作为多种恶性肿瘤的治疗靶点。本研究结果表明,INPP4B的表达水平在胰腺癌耐药细胞/组织中显著高于未耐药组,进一步的生存分析显示INPP4B低表达水平的患者相比高表达水平者有着更好的预后。结合既往研究,我们猜测INPP4B与胰腺癌吉西他滨耐药之间可能通过磷酸化/去磷酸化代谢产生关联,考虑到INPP4B主要的功能是将PI(3,4)P2去磷酸化生成PI(3)P,而吉西他滨发挥作用同样需在细胞中经历两步关键的磷酸化反应,磷酸基(P)作为其中共同的代谢产物可能是它们之间关联的核心,而其中的具体机制有待后续的体内、外实验进一步去探索。
INPP4B作为一种化疗响应的标志物已有报道。Asleh等[28]针对转移性乳腺癌患者为期13年的中位随访发现,被定义为“巢蛋白+或INPP4B-”的乳腺癌患者在接受含吉西他滨的化疗时,OS率明显优于多西他赛单药治疗(HR=0.31,95% CI:0.16~0.60)。这一研究结论从另一角度提示INPP4B阳性的转移性乳腺癌患者可能与不佳的吉西他滨治疗响应相关,与我们在胰腺癌患者中的发现不谋而合,尽管该结论仍需要通过更大的前瞻性临床研究进行验证。
总之,本研究发现,在吉西他滨耐药胰腺癌细胞株Mia GR与亲本细胞系Mia WT之间的所有差异表达基因中,INPP4B很可能是与耐药现象有关联的关键生物标志物,同时INPP4B在耐药/未耐药胰腺癌患者组织及正常胰腺组织间表现出显著的表达水平差异,且与这些患者的预后显著相关。因此,后续我们计划以INPP4B分子相关的磷酸化/去磷酸化代谢为基础,深入挖掘其在胰腺癌吉西他滨耐药中的调控靶点及作用机制,为后续开发新的靶向治疗药物以改善胰腺癌吉西他滨化疗现状奠定基础。
致谢:
作者感谢西安交通大学第一附属医院生物样本信息资源中心工作人员及病理科临床医师协助进行病理组织获取及病理学评估。
利益冲突声明:
所有作者均声明不存在利益冲突。
作者贡献声明:
任加强收集数据和病理学标本,完成了大部分实验,分析数据并进行可视化分析,撰写了初稿;武帅参与数据收集和部分实验,提供思路和技术指导;苏童协助完成数据分析和可视化分析;李杰和韩亮参与实验思路和技术指导,修改初稿;仵正监督实验并完成初稿的最终修订和审阅,负责投稿事宜。所有作者都阅读并通过了最终稿件。
[参考文献]
[1] SIEGEL R, MILLER K D, WAGLE N S, et al. Cancer statistics, 2023[J]. CA A Cancer J Clin, 2023, 73: 17-48.
[2] MOORE A, DONAHUE T. Pancreatic cancer[J]. JAMA, 2019, 322(14): 1426.
[3] ZHANG L F, WU J H, LING M T, et al. The role of the PI3K/ Akt/mTOR signalling pathway in human cancers induced by infection with human papillomaviruses[J]. Mol Cancer, 2015, 14: 87.
[4] TEMPERO M A, MALAFA M P, AL-HAWARY M, et al. Pancreatic adenocarcinoma, version 2.2017, NCCN clinical practice guidelines in oncology[J]. J Natl Compr Canc Netw, 2017, 15(8): 1028-1061.
[5] VO T T T, FRUMAN D A. INPP4B is a tumor suppressor in the context of PTEN deficiency[J]. Cancer Discov, 2015, 5(7): 697-700.
[6] GEWINNER C, WANG Z C, RICHARDSON A, et al. Evidence that inositol polyphosphate 4-phosphatase type Ⅱ is a tumor suppressor that inhibits PI3K signaling[J]. Cancer Cell, 2009, 16(2): 115-125.
[7] DZNELADZE I, WOOLLEY J F, ROSSELL C, et al. SubID, a non-median dichotomization tool for heterogeneous populations, reveals the pan-cancer significance of INPP4B and its regulation by EVI1 in AML[J]. PLoS One, 2018, 13(2): e0191510.
[8] ZHAI S Y, LIU Y B, LU X X, et al. INPP4B as a prognostic and diagnostic marker regulates cell growth of pancreatic cancer via activating AKT[J]. Onco Targets Ther, 2019, 12: 8287-8299.
[9] ZHANG B, WANG W D, LI C H, et al. Inositol polyphosphate- 4-phosphatase type Ⅱ plays critical roles in the modulation of cadherin-mediated adhesion dynamics of pancreatic ductal adenocarcinomas[J]. Cell Adh Migr, 2018, 12(6): 548-563.
[10] FARRELL A S, JOLY M M, ALLEN-PETERSEN B L, et al. MYC regulates ductal-neuroendocrine lineage plasticity in pancreatic ductal adenocarcinoma associated with poor outcome and chemoresistance[J]. Nat Commun, 2017, 8(1): 1728.
[11] SHAO F, HUANG M, MENG F T, et al. Circular RNA signature predicts gemcitabine resistance of pancreatic ductal adenocarcinoma[J]. Front Pharmacol, 2018, 9: 584.
[12] SAIKI Y, YOSHINO Y, FUJIMURA H, et al. DCK is frequently inactivated in acquired gemcitabine-resistant human cancer cells[J]. Biochem Biophys Res Commun, 2012, 421(1): 98-104.
[13] ZHOU J R, ZHANG L S, ZHENG H L, et al. Identification of chemoresistance-related mRNAs based on gemcitabineresistant pancreatic cancer cell lines[J]. Cancer Med, 2020, 9(3): 1115-1130.
[14] SCHWARTZ L H, LITIÈRE S, DE VRIES E, et al. RECIST 1.1-Update and clarification: from the RECIST committee[J]. Eur J Cancer, 2016, 62: 132-137.
[15] ZHANG X, ZHENG S Y, HU C H, et al. Cancer-associated fibroblast-induced lncRNA UPK1A-AS1 confers platinum resistance in pancreatic cancer via efficient double-strand break repair[J]. Oncogene, 2022, 41(16): 2372-2389.
[16] BILIMORIA K Y, BENTREM D J, KO C Y, et al. Validation of the 6th edition AJCC pancreatic cancer staging system: report from the National Cancer Database[J]. Cancer, 2007, 110(4): 738-744.
[17] RODGERS S J, OOMS L M, OORSCHOT V M J, et al. INPP4B promotes PI3Kα-dependent late endosome formation and Wnt/ β-catenin signaling in breast cancer[J]. Nat Commun, 2021, 12(1): 3140.
[18] CHEN Y, SUN Z Y, QI M, et al. INPP4B restrains cell proliferation and metastasis via regulation of the PI3K/AKT/ SGK pathway[J]. J Cell Mol Med, 2018, 22(5): 2935-2943.
[19] TEMPERO M A, MALAFA M P, AL-HAWARY M, et al. Pancreatic adenocarcinoma, version 2.2021, NCCN clinical practice guidelines in oncology[J]. J Natl Compr Canc Netw, 2021, 19(4): 439-457.
[20] GROOT V P, REZAEE N, WU W C, et al. Patterns, timing, and predictors of recurrence following pancreatectomy for pancreatic ductal adenocarcinoma[J]. Ann Surg, 2018, 267(5): 936-945.
[21] KANDA M, MATTHAEI H, WU J, et al. Presence of somatic mutations in most early-stage pancreatic intraepithelial neoplasia[J]. Gastroenterology, 2012, 142(4): 730-733.e9.
[22] WADDELL N, PAJIC M, PATCH A M, et al. Whole genomes redefine the mutational landscape of pancreatic cancer[J]. Nature, 2015, 518(7540): 495-501.
[23] DAUER P, NOMURA A, SALUJA A, et al. Microenvironment in determining chemo-resistance in pancreatic cancer: neighborhood matters[J]. Pancreatology, 2017, 17(1): 7-12.
[24] BEATTY G L, EGHBALI S, KIM R. Deploying immunotherapy in pancreatic cancer: defining mechanisms of response and resistance[J]. Am Soc Clin Oncol Educ Book, 2017, 37: 267-278.
[25] GOLAN T, HAMMEL P, RENI M, et al. Maintenance olaparib for germline BRCA-mutated metastatic pancreatic cancer[J]. N Engl J Med, 2019, 381(4): 317-327.
[26] PISHVAIAN M J, BLAIS E M, BRODY J R, et al. Overall survival in patients with pancreatic cancer receiving matched therapies following molecular profiling: a retrospective analysis of the Know Your Tumor registry trial[J]. Lancet Oncol, 2020, 21(4): 508-518.
[27] MINI E, NOBILI S, CACIAGLI B, et al. Cellular pharmacology of gemcitabine[J]. Ann Oncol, 2006, 17(Suppl 5): v7-v12.
[28] ASLEH K, LYCK CARSTENSEN S, TYKJAER JØRGENSEN C L, et al. Basal biomarkers nestin and INPP4B predict gemcitabine benefit in metastatic breast cancer: samples from the phase Ⅲ SBG0102 clinical trial[J]. Int J Cancer, 2019, 144(10): 2578-2586.


