Nature Genetics:从基因剪接到精准医疗:复杂疾病的新靶点
时间:2024-12-05 14:02:56 热度:37.1℃ 作者:网络
引言
近年来,单细胞RNA测序技术(single-cell RNA sequencing, scRNA-seq)的发展为探索复杂疾病背后的遗传和分子机制提供了全新视角。传统的基因组广泛关联研究(GWAS)虽然发现了许多与复杂性状相关的基因位点,但大多数位点位于非编码区域,其功能机制至今仍未完全解明。此外,研究还发现,不同细胞类型和环境状态对基因表达的调控具有高度特异性,这使得仅基于总体数据的研究难以揭示细胞类型特异性调控的复杂性。
12月3日Nature Genetics的研究报道“Single-cell RNA sequencing of peripheral blood links cell-type-specific regulation of splicing to autoimmune and inflammatory diseases”,以单细胞RNA测序技术为核心,聚焦于约100万来自474名东亚、东南亚及南亚供体的外周血单个核细胞(PBMC),首次在大规模亚洲人群中对可变剪接(alternative splicing, AS)进行深入解析。研究特别关注剪接数量性状位点(splicing quantitative trait loci, sQTL),揭示了其在细胞类型、性别及遗传背景上的差异性。同时,通过结合GWAS数据,研究发现了这些sQTL在免疫相关性疾病遗传风险中的重要作用,并验证了一些可能影响疾病风险的亚洲特异性剪接位点。例如,研究发现一个与Graves病风险相关的sQTL,通过破坏TCHP基因外显子4的剪接位点,影响了东亚人群中该疾病的遗传易感性。
该研究的亮点在于创新性地利用5′端测序策略和随机mRNA断裂及修复机制,不仅提高了单细胞数据的外显子覆盖率,还揭示了多个细胞类型的剪接调控模式。通过结合单细胞水平的基因组和表型数据,这项研究为解析复杂疾病的遗传机制提供了新的方法学框架,也为探索多样化遗传背景对疾病风险的影响提供了重要的科学依据。这些发现不仅有助于深化我们对基因剪接调控的理解,也为开发精准医疗策略奠定了基础。

揭秘生命的分子拼图:可变剪接与复杂疾病的关联
在我们身体的每一个细胞中,基因就像一本密码书,决定了生命的基本构造与功能。而这本书并不是每一页都直接使用,有时甚至会经过复杂的“剪裁”和“拼接”,形成特定的表达内容——这就是可变剪接(Alternative Splicing, AS)。它是生命分子层面的“拼图大师”,可以通过调控单一基因产生多种不同的蛋白质,从而显著扩展基因组的功能复杂性。
研究表明,可变剪接在许多复杂性状和疾病中扮演着至关重要的角色。特别是在免疫系统中,细胞在应对病原体的过程中需要灵活调整自己的分子机制,而可变剪接恰好能满足这一需求。然而,这种灵活性也可能成为“双刃剑”,如果剪接调控出现异常,可能会导致多种疾病,包括自身免疫疾病、炎症性疾病甚至癌症。
这项研究通过单细胞RNA测序(scRNA-seq)技术,对近100万外周血单个核细胞(PBMC)进行了深入分析,涵盖474名不同亚洲遗传背景的供体,首次揭示了剪接调控的细胞类型特异性、性别差异及祖源依赖性。结果显示,在48个性别偏倚的剪接事件和1031个祖源偏倚的事件中,大部分与细胞特异性调控密切相关。例如,研究发现了一种在东亚人群中特有的剪接数量性状位点(splicing quantitative trait loci, sQTL),其破坏了TCHP基因外显子4的剪接位点,显著增加了Graves病的遗传风险。
亚洲人群的独特基因奥秘:多样化的研究背景
亚洲是全球遗传多样性的重要宝库,其广袤的地域和悠久的历史孕育了丰富的基因变异。然而,遗憾的是,传统的基因组研究长期以欧美人群为主,导致许多与亚洲特定人群相关的重要遗传信息被忽视。而这项研究正是通过聚焦亚洲遗传背景,揭示了隐藏在这些基因多样性背后的奥秘。
该研究依托于“亚洲免疫多样性图谱”(Asian Immune Diversity Atlas, AIDA),汇集了来自东亚(日本、韩国)、东南亚(新加坡华人和马来人)及南亚(新加坡印度人)474名供体的样本,覆盖了超过100万个外周血单个核细胞(PBMC)。这一多样化的研究人群不仅展现了亚洲人群的基因复杂性,也为探索特定人群中的遗传变异和疾病风险提供了重要机会。研究发现,约1031个剪接事件具有明显的祖源偏倚,例如SPSB2基因的一个重要单核苷酸多态性(SNP)位点rs11064437,其小等位基因频率(MAF)在东亚人群中高达30.7%,而在南亚人群中仅为3.4%。这一差异显然与遗传背景密切相关。
此外,研究还发现了一个具有东亚特异性的剪接数量性状位点(sQTL),与Graves病的风险显著相关。这个位点通过破坏TCHP基因外显子4的剪接位点,可能导致东亚人群中该疾病的高发。这些发现不仅展示了亚洲遗传多样性的科学价值,也提醒我们,特定人群的遗传背景可能对疾病的发生机制有深远影响。
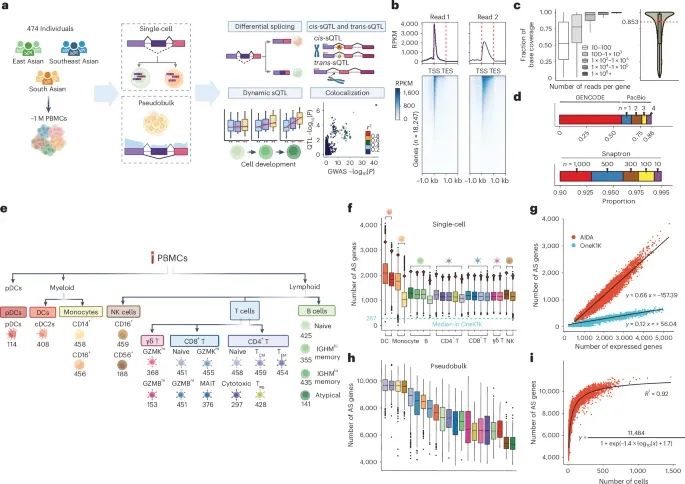
通过大规模5′端单细胞RNA测序(5′ scRNA-seq)对外周血单个核细胞(PBMC)中21种细胞类型和可变剪接基因进行分析(Credit: Nature Genetics)
研究设计与样本规模
图a描述了AIDA研究的设计,覆盖474名东亚、东南亚和南亚供体,总计约100万个单细胞样本。这些样本被用来探索不同细胞类型中的可变剪接和剪接数量性状位点(sQTL)。
数据读取覆盖情况
图b通过图形和热图展示了5′端测序数据的覆盖特性。研究发现,读取1(read 1)更倾向于转录起始位点(TSS),而读取2(read 2)更均匀地分布于基因主体。这种分布特点提升了外显子的覆盖率,为研究可变剪接提供了更高质量的数据。
基因覆盖率与读取深度的关系
图c展示了每个基因的覆盖率随读取深度的增加而提升。在所有表达基因中,中位数的外显子覆盖率达到85.3%,表明测序数据具有高覆盖质量。这些结果为剪接分析提供了可靠的基础。
剪接事件的验证与稳定性
图d展示了LeafCutter剪接事件的验证结果。59.3%的剪接事件在GENCODE中有注释,85.9%在PacBio长读测序中得到了验证。超过93%的检测到的剪接位点在超过1,000个样本中存在,进一步说明了数据的可靠性。
PBMC亚型的分析与细胞类型数量
图e对21种PBMC亚型进行了深入分析,细胞类型按照血液生成系统的谱系进行分类。不同细胞类型中剪接分析和sQTL分析的样本量得到了详细列举。
单细胞水平的剪接基因数量
图f显示了在单细胞水平上每种细胞类型中检测到的可变剪接基因数量(NODGs),其中红色菱形表示每个细胞中检测到的基因的平均数量。与OneK1K数据集相比,AIDA数据集检测到的剪接基因数量显著增加。
剪接基因数量的相关性与规模效应
图g指出,可变剪接基因的数量与表达基因数量正相关,AIDA数据集中这种相关性显著高于OneK1K数据集。
伪整体水平的剪接基因检测
图h和图i展示了在伪整体水平(pseudobulk)分析中检测到的剪接基因数量,随着细胞数量的增加而呈现饱和趋势,达到约11,500个基因。
从单细胞到多维数据:技术创新如何改变研究范式
传统的基因组学技术通常基于整个人体组织的平均表达水平,这种方法虽然提供了宏观视角,但往往掩盖了不同细胞类型间的关键差异。而单细胞RNA测序(scRNA-seq)的出现,犹如一把放大镜,让研究人员能够以单细胞为单位精确解析基因表达和调控过程,从而揭示疾病背后的微观机制。
该研究采用了单细胞5′端RNA测序技术,并结合随机mRNA断裂和修复机制,大幅提升了数据的外显子覆盖率。研究团队对474名亚洲供体的约100万个外周血单个核细胞(PBMC)进行了测序,覆盖了21种细胞类型,检测到了超过11500个可变剪接基因,是以往技术的4倍以上。特别值得注意的是,这种方法成功突破了传统单细胞技术的局限性,能够更全面地解析基因的可变剪接模式。
例如,研究通过单细胞数据发现了与T细胞分化密切相关的PTPRC基因(编码CD45蛋白)的不同剪接形式。在幼稚T细胞中,主要表达包含外显子4、5和6的CD45RA+形式,而在激活和记忆T细胞中,则以跳过这些外显子的CD45RO形式为主。这种剪接模式的动态变化对免疫应答的调控至关重要,展示了单细胞技术在捕捉细胞分化和功能转变中的独特优势。
更重要的是,单细胞RNA测序还实现了在个体、细胞类型及基因调控层面上的多维数据整合。例如,研究通过剪接数量性状位点(sQTL)分析,揭示了细胞特异性基因剪接与复杂疾病之间的关联。
剪接调控的密码:剪接数量性状位点 (sQTL) 的发现
基因剪接是一种精密的“分子编辑”,决定着基因如何表达成特定功能的蛋白质。而在这种编辑过程中,剪接数量性状位点(splicing quantitative trait loci, sQTL)则如同隐藏的调控开关,精确地调节基因的剪接模式。研究发现,sQTL广泛存在于人类基因组中,但其细胞特异性和遗传背景的影响尚未被充分探索。该研究首次以大规模亚洲人群为样本,深入解析了sQTL在复杂疾病遗传风险中的作用。
通过单细胞RNA测序技术,研究团队鉴定出了10,874个蛋白编码基因和703个长链非编码RNA的sQTL,并揭示了许多具有细胞类型特异性或性别差异的调控模式。研究的在T细胞中特异调控的sQTL,该位点位于HNRNPLL基因中,并通过影响PTPRC(编码CD45蛋白)的剪接,调节T细胞活化和分化的关键过程。此外,研究发现了865个在B细胞分化过程中呈动态变化的剪接事件,表明sQTL的调控效应在细胞发育过程中可能发生显著改变。
研究还揭示了性别和祖源对sQTL的重要影响。性别偏倚的剪接事件中,FLNA基因的一个短型剪接形式在女性供体的成熟T细胞和自然杀伤细胞中显著增加,与类风湿关节炎的免疫调控机制可能有关。而祖源相关的sQTL分析则发现,SPSB2基因的一个位点在东亚、东南亚和南亚人群中的等位基因频率差异可能引发不同的剪接结果,从而影响该基因的功能。
免疫疾病的基因钥匙:从遗传调控到精准医疗
免疫系统是保护人体免受病原体侵害的坚强防线,但遗传调控的失误可能让它成为疾病的推手。近年来,研究发现,与免疫相关的基因调控异常,如可变剪接调控的紊乱,可能是许多自身免疫疾病的关键致病因素。
在这项研究中,研究团队通过单细胞RNA测序技术,鉴定出了多个与免疫相关性疾病密切相关的sQTL,尤其是在亚洲特有的遗传背景中。一个特别重要的发现是与Graves病相关的sQTL。这一sQTL位于TCHP基因,破坏了其外显子4的剪接位点,从而改变了基因表达模式。通过功能验证,研究证实这一遗传变异显著增加了东亚人群中患Graves病的风险,这为解析疾病的分子机制提供了新的线索。
此外,研究还揭示了HNRNPLL和PTPRC基因之间的剪接调控网络。HNRNPLL基因的表达变化通过调控PTPRC(编码CD45蛋白)的剪接形式,直接影响T细胞的发育和功能,这一机制可能与多种免疫相关疾病的发生密切相关。
通过结合单细胞数据和GWAS分析,研究还发现了多种免疫疾病(如系统性红斑狼疮和类风湿关节炎)与sQTL的显著关联。这些发现不仅深化了我们对免疫疾病遗传基础的理解,也为精准医疗的实现提供了潜力巨大的新靶点。
性别与祖源的影响:基因剪接如何反映多样化的调控
人类基因组的多样性不仅表现在个体差异,也深深受到性别和祖源的影响。在基因表达调控中,这种差异尤为显著,特别是在可变剪接(Alternative Splicing, AS)的调控机制中。研究发现,性别和祖源相关的遗传变异可以直接影响基因剪接的方式,从而塑造不同个体对环境和疾病的独特适应性。
该研究通过单细胞RNA测序,揭示了48个性别偏倚的差异剪接事件(DSEs),它们影响了32个基因的表达。例如,FLNA基因的一个短型剪接形式(ENST00000498491)在女性成熟T细胞和自然杀伤细胞中显著增多,这种性别差异可能与女性在类风湿关节炎等自身免疫疾病中的高发率相关。此外,在免疫相关基因PTPRC(编码CD45蛋白)的剪接中,也观察到了性别特异性调控的趋势,这进一步说明了性别在免疫调控中的重要性。
除了性别差异,祖源相关的遗传变异也对基因剪接产生了深远影响。在该研究中,SPSB2基因的一个剪接位点因祖源差异而具有显著的等位基因频率变化。例如,位点rs11064437在东亚人群中的小等位基因频率为30.7%,而在南亚人群中仅为3.4%。这一差异通过影响SPSB2外显子2的剪接效率,可能导致不同人群在免疫反应和疾病易感性上的显著差异。
从B细胞发育看基因剪接的动态变化
B细胞是免疫系统的重要组成部分,其发育过程如同精密的“分子进化”,由幼稚B细胞逐步分化为记忆B细胞。这一过程不仅伴随着基因表达的变化,更展现了复杂的动态剪接模式,为B细胞适应多样化的免疫需求提供了分子基础。
该研究深入解析了B细胞在发育过程中的剪接动态变化。研究以52,964个B细胞为对象,按分化轨迹划分为幼稚B细胞、IGHMhi记忆B细胞和IGHMlo记忆B细胞三种主要亚群。数据分析显示,865个内含子剪接事件的使用与B细胞的发育轨迹显著相关,呈现出三种独特的动态模式:阶梯式变化、线性变化和抛物线变化。
阶梯式变化的典型例子是PAX5基因,这是一个B细胞特异的转录因子,对B细胞的命运决定至关重要。研究发现,在幼稚B细胞中,PAX5A剪接形式占主导地位,而随着B细胞分化为记忆B细胞,PAX5B剪接形式逐渐增加。这种剪接模式的改变可能反映了PAX5在不同发育阶段对基因表达的调控差异。
线性变化的模式则出现在PTPRC基因(编码CD45蛋白)中,其外显子4的使用率在B细胞从幼稚状态向记忆状态转变时逐步降低。这种变化可能影响B细胞对抗原的识别和响应能力,是B细胞成熟的重要分子标志。
抛物线变化的一个例子是DOCK8基因,其外显子1的剪接使用在记忆B细胞的中间发育阶段达到峰值,随后下降。这种变化模式可能反映了DOCK8在特定发育阶段的瞬时功能需求。
这些动态剪接模式不仅揭示了基因表达在B细胞发育过程中的复杂调控,还为理解免疫系统的多样性提供了重要线索。
研究中的每一个数据点,都可能是解开疾病谜题的关键拼图。该研究以单细胞RNA测序为核心,结合剪接数量性状位点(sQTL)的深度挖掘,为解析复杂疾病的遗传和分子机制提供了新的方向。这些发现不仅扩展了基础研究的边界,也为精准医疗带来了前所未有的希望。
参考文献
Tian, C., Zhang, Y., Tong, Y. et al. Single-cell RNA sequencing of peripheral blood links cell-type-specific regulation of splicing to autoimmune and inflammatory diseases. Nat Genet (2024). https://doi.org/10.1038/s41588-024-02019-8


