Cell:肿瘤免疫疗法会自我限制其疗效,通过激活Treg细胞,降低免疫治疗效果
时间:2021-06-28 10:01:36 热度:37.1℃ 作者:网络
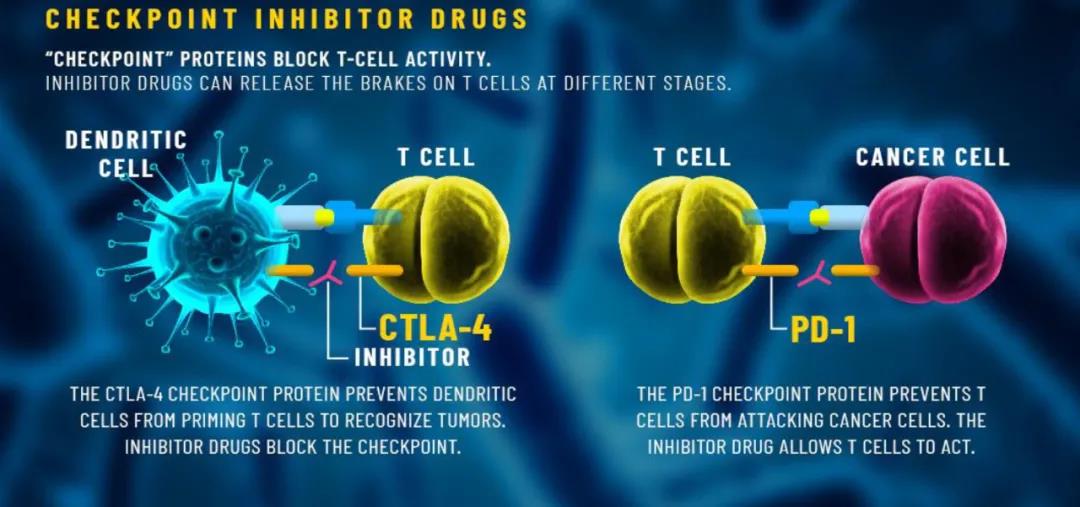
随着肿瘤免疫治疗逐渐成为临床靶向药物研究开发的重要方向之一,越来越多的肿瘤免疫治疗基因靶点进入研发视野。PD-1和PD-L1靶点无疑是当前最热门的焦点。大量已经上市和正在研究的PD-1 / PD-L1抑制剂取得了良好的临床疗效。
除了PD-1 / PD-L1以外,CTLA-4也同样属于热门肿瘤免疫治疗靶点。目前临床应用的CTLA-4抑制剂,包括百时美施贵宝公司的伊匹单抗(Ipilimumab)和阿斯利康公司的曲美木单抗(Tremelimumab),已广泛应用于肿瘤免疫治疗,积累了大量的临床应用经验。
CTLA-4,即细胞毒性T淋巴细胞相关蛋白4,是一种白细胞分化抗原,是T细胞上的一种跨膜受体,其作为免疫检查点起作用并下调免疫应答。CTLA-4通过与抗原细胞表面的受体结合,从而起到开关作用,终止免疫反应。而CTLA-4抑制剂通过抑制CTLA-4分子,则能使T细胞大量增殖、攻击肿瘤细胞。
然而,免疫检查点只是多层次调节机制系统的一个组成部分。调节性T细胞(Treg)的活性构成了另一层免疫调节,但它们的免疫抑制功能如何在肿瘤微环境(TME)中受到调节仍然未知。
近日,哈佛大学医学院的研究团队在国际顶尖学术期刊 Cell 发表了题为:Expansion of tumor-associated Treg cells upon disruption of a CTLA-4-dependent feedback loop 的研究论文。
该研究使用活体成像技术发现,抗癌药物抑制CTLA-4分子并激活CD8和CD4效应T细胞,从而杀死癌症。然而,CTLA-4 抑制还会导致Treg细胞的扩增,从而降低免疫疗法的效果。
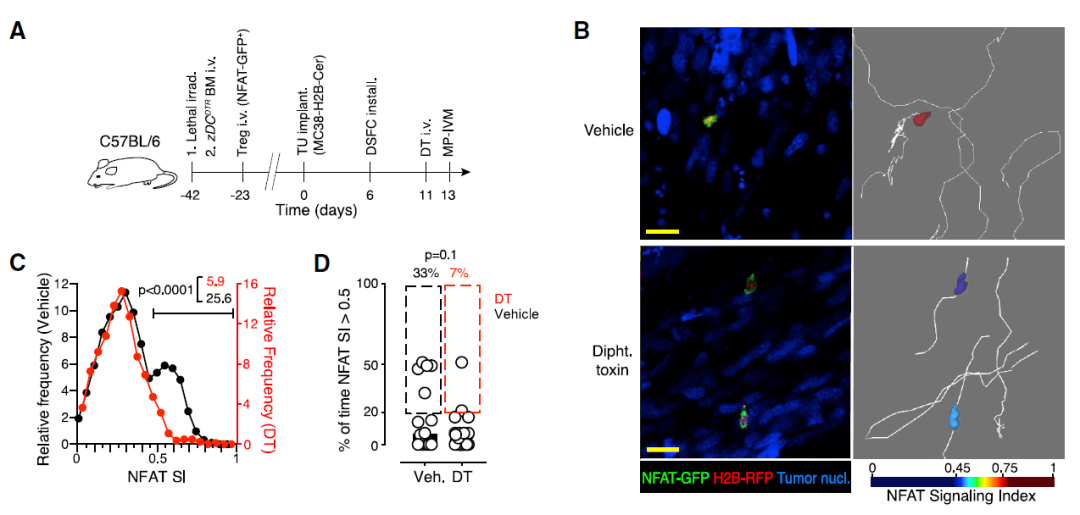
为了通过体内成像检查肿瘤组织中Treg细胞活化的单细胞动力学,研究团队用表达NFAT-GFP和荧光组蛋白融合蛋白H2B-RFP的逆转录病毒感染Treg细胞并将其植入小鼠皮下。
结果显示,与传统的辅助性Th细胞不同,这些Treg细胞在稳定的抗原接触过程中被激活,表明Treg细胞库在TME中被预先富集以识别抗原,而Th细胞库中只有罕见的细胞识别抗原。
另外,尽管Treg细胞仅部分稳定了它们与肿瘤组织中抗原提呈细胞(APC)的相互作用,但这些不稳定的相互作用触发了激活NFAT通路的T细胞抗原受体(TCR)信号。
因为常规树突状细胞(cDC)有助于招募和激活肿瘤浸润性细胞毒性T淋巴细胞(CTL),研究团队假设它们也可能激活Treg细胞。因此,他们构建了zDCDTR/B6照射骨髓嵌合体(BMC)。cDC急性消融后Treg细胞的迁移不受限制,但活化频率显着下降,表明cDC在局部Treg细胞活化中发挥重要作用。
考虑到肿瘤浸润Th和Treg细胞的TCR序列在很大程度上是不同的,Treg细胞会破坏肿瘤内APC与Th细胞的接触。因此,研究团队比较了表达相同TCR的Treg和Th细胞的行为。
结果提示,Treg和Th细胞与APC相互作用的差异不是由TCR亲和力的内在差异所驱动,而是由Treg细胞引入TME的变化驱动的。这些变化可能包括APC功能的改变,导致APC不仅与Th接触,而且与Treg细胞接触不稳定。
那么,Treg细胞是如何影响Th细胞的稳定性及其与肿瘤相关APC的相互作用的呢?
因为Treg细胞可以利用共刺激B7家族蛋白的CTLA-4减少SLO中APC上的CD80和CD86的数量,并且考虑到肿瘤浸润性Treg细胞上CTLA-4的高表达,研究团队探索了CTLA-4调节肿瘤组织中T细胞与APC相互作用稳定性的功能。
他们发现CTLA-4限制了CD80和CD86在肿瘤浸润cDC上的表达,限制了局部Treg细胞的增殖,并且需要伴随的Treg细胞失活来实现肿瘤排斥。
因此,Treg 细胞通过依赖于CTLA-4和CD28的反馈回路进行自我调节,该回路根据局部共刺激的数量调整其群体大小。它通过CTLA-4抑制的破坏可能会抵消癌症患者的治疗益处。

综上,该研究在临床前肿瘤模型中证实,Treg细胞在常规树突状细胞(cDC)上表达共刺激蛋白CD80和CD86。Treg细胞通过利用其对CD28共刺激信号的依赖性来不断调整其群体大小,以达到耗尽CD28配体CD80和CD86的目的。由此产生的平衡状态被CTLA-4抑制剂所打破,这会产生肿瘤内过度增殖的Treg细胞,使得患者处于免疫抑制状态并持续对抗免疫介导的肿瘤排斥,这可能会抵消癌症患者的抗CTLA-4治疗的疗效。
原始出处:
Francesco Marangoni, et al. Expansion of tumor-associated Treg cells upon disruption of a CTLA-4-dependent feedback loop. Cell, 2021. DOI:https://doi.org/10.1016/j.cell.2021.05.027.


