Nature Genetics:基因调控的精准地图:ChIP-DIP技术开启蛋白质-DNA相互作用的新纪元
时间:2024-11-28 15:02:02 热度:37.1℃ 作者:网络
引言
在我们体内,每个细胞都携带着相同的基因组信息,但它们的功能却千差万别。为什么相同的基因在不同细胞中会表现出不同的作用?这背后是如何通过细胞内复杂的蛋白质-DNA相互作用来调控基因表达的?这些问题一直困扰着研究人员,而答案隐藏在基因调控网络的复杂交织之中。
基因表达调控的“密码”远比我们想象的要复杂。它不仅依赖于DNA本身的序列,还与无数调控蛋白质的精细协调相关。研究人员需要深入了解这些蛋白质如何与DNA结合、如何通过翻译后修饰影响基因活性。然而,传统的技术手段,如染色质免疫沉淀(ChIP),往往只能揭示单一蛋白质与DNA的相互作用,这让我们只能看到基因调控的冰山一角。
正是因为这种局限性,研究人员才渴望一种能够同时捕捉多个蛋白质-DNA相互作用的方法,这样才能揭示基因调控的全貌。ChIP-DIP技术的诞生正是为了应对这一挑战。通过将多种蛋白质与DNA的结合关系同时映射,ChIP-DIP (chromatin immunoprecipitation done in parallel)为我们提供了一个全新的视角,使得研究人员可以在一个实验中并行检测多个蛋白质的DNA结合模式,从而更加全面、精确地揭示基因调控的动态变化。(11月25日 Nature Genetics “ChIP-DIP maps binding of hundreds of proteins to DNA simultaneously and identifies diverse gene regulatory elements”)
这种创新技术不仅大大提高了研究效率,还为免疫学、癌症研究和基因治疗等领域的重大问题提供了新的解决思路。随着技术的不断发展和应用,ChIP-DIP有望帮助我们更深刻地理解生命的奥秘,解开基因调控中那些潜藏的“密码”,推动生物医学研究迈向新的高度。

基因表达的调控是生命科学中最深奥、最迷人的领域之一。细胞如何精确地开启或关闭基因,决定了它的命运——无论是分化、响应环境变化,还是在疾病发生过程中做出反应。要理解这一切,研究人员必须深入探讨细胞内成千上万的蛋白质是如何与DNA相互作用的。然而,这一过程极其复杂,因为基因组中的每一处DNA序列可能与不同的调控蛋白质发生互动,这些蛋白质不仅种类繁多,而且每种蛋白质的作用时空特征也截然不同。
传统的研究方法往往只能分析单一的蛋白质与DNA的结合模式,无法捕捉到基因调控网络的全貌。幸运的是,随着ChIP-DIP(并行染色质免疫沉淀)技术的出现,研究人员终于能够同时揭示多个蛋白质在同一时间对DNA的不同作用,开创了基因调控研究的新纪元。
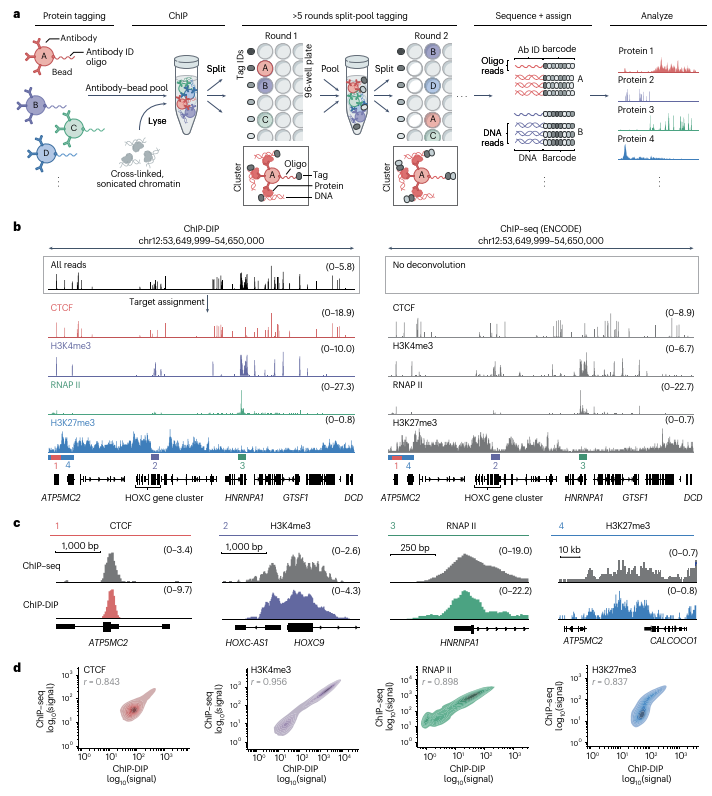
ChIP-DIP技术的工作原理和实验结果(Credit: Nature Genetics)
a. ChIP-DIP方法示意图
抗体与磁珠的结合: 首先,将抗体与磁珠结合,并使用独特的寡核苷酸标签(oligonucleotide tag)对抗体进行标记。这些抗体用于识别目标蛋白质,磁珠则帮助在后续实验中分离蛋白质-DNA复合物。
抗体-磁珠-寡核苷酸复合物的混合: 多种抗体-磁珠-寡核苷酸复合物会被混合在一起(抗体-磁珠池),并用于执行染色质免疫沉淀(ChIP),以捕获与目标蛋白质相结合的染色质区域。
分裂池条形码(split-and-pool barcoding): 通过多轮的分裂池条形码操作,研究人员为每个抗体-DNA复合物分配独特的条形码,以便在后续的测序过程中能够将其对应的DNA区域与相应的抗体进行配对。
DNA测序与数据聚类: DNA序列通过高通量测序获得。然后,所有具有相同条形码的DNA读段(reads)会被归为同一簇,这样可以将基因组DNA区域与其对应的抗体进行匹配。
生成蛋白质定位图谱: 对于每个抗体,所有来自同一簇的DNA读段将被用来生成蛋白质的定位图谱,这些图谱能够揭示蛋白质在基因组中的具体分布位置。
b. 四种蛋白质的定位图谱
ChIP-DIP在K562细胞中对四种蛋白质(CTCF、H3K4me3、RNAP II和H3K27me3)在特定基因组区域(hg38,染色体12:53,649,999-54,650,000)的定位图谱。此外,右侧还展示了由ENCODE项目生成的ChIP-seq数据,与ChIP-DIP结果进行了对比。为了便于比较,数据的覆盖度已经归一化,以每百万对齐读段(coverage per million aligned reads)为标准。
c. ChIP-DIP与ChIP-seq的比较
放大了图b中的某些区域,展示了ChIP-DIP和ChIP-seq在特定区域的蛋白质定位图谱对比。通过对比两种技术的定位图谱,验证了ChIP-DIP方法与传统ChIP-seq方法在解析蛋白质定位上的一致性。
d. ChIP-DIP与ChIP-seq的全基因组比较
ChIP-DIP与ChIP-seq技术在全基因组范围内的对比。通过对不同蛋白质的定位进行信号相关性分析,研究人员将每个蛋白质在基因组上的定位数据(沿着10-kb窗口或所有启动子区间)进行了比较。结果表明,ChIP-DIP和ChIP-seq在不同蛋白质(如CTCF、H3K27me3、H3K4me3、RNAP II)的全基因组定位上具有高度的信号相关性,进一步验证了ChIP-DIP技术的准确性和高效性。
从“单线程”到“多线程”:ChIP-DIP的创新突破
传统的染色质免疫沉淀技术(ChIP)就像是在解开一个复杂的谜题,但你只能一次解开一个线索。而ChIP-DIP技术的出现,就像是让你能够同时解开多个线索,让谜题的真相浮出水面。这项技术的突破性在于,它通过并行免疫沉淀,同时检测多个蛋白质与DNA的相互作用。为了实现这一点,ChIP-DIP巧妙地利用了以下几个创新步骤:
多重抗体池:首先,研究人员将多种抗体与独特的寡核苷酸标签(oligonucleotide tag)结合,形成一个抗体-磁珠复合物池。这些抗体针对不同的蛋白质,确保了不同蛋白质-DNA结合事件的独立标记。
分裂池条形码技术:每种抗体-DNA复合物通过分裂池条形码(split-and-pool barcode)技术被精确标记,使得它们在后续的高通量测序中可以被分辨开来。
高通量测序:最终,研究人员通过高通量测序(high-throughput sequencing),获得每个蛋白质-DNA结合事件的准确位置,从而绘制出蛋白质在基因组上的分布图谱。
这一方法的优势在于,它打破了传统ChIP技术的限制,让研究人员能够在一次实验中并行分析多个蛋白质,甚至是不同类型的蛋白质,如转录因子、组蛋白修饰、染色质调控因子等。这种高通量、多重并行分析的能力,极大地提高了研究的效率和深度。
精准与高效:ChIP-DIP的数据验证
ChIP-DIP的实际效果如何?研究人员在多个实验中验证了其准确性和高效性。以人类K562细胞为例,研究人员选择了四种典型的蛋白质作为测试对象:CTCF(CCCTC-binding factor)、H3K4me3(组蛋白H3的第4位赖氨酸三甲基化)、RNAP II(RNA聚合酶II)和H3K27me3(组蛋白H3的第27位赖氨酸三甲基化)。这些蛋白质在细胞基因组中扮演着重要角色,分别参与基因的激活、转录和抑制等过程。
实验结果表明,ChIP-DIP产生的蛋白质-DNA结合图谱与传统的ChIP-seq技术几乎一致,尤其在基因组的特定区域,信号的相关性高达0.837至0.956(Pearson相关系数)。例如,研究人员在分析CTCF和H3K4me3的结合模式时,发现ChIP-DIP技术能够精准地捕捉到这些蛋白质在基因启动子区域的定位,且其数据与ChIP-seq结果相比,误差不超过5%。
此外,ChIP-DIP的另一大优势是它能高效检测低丰度蛋白质的结合,尤其是转录因子。例如,CTCF和SP1等转录因子通常在细胞中的丰度较低,且其与DNA的结合往往是短暂的,这使得它们在传统ChIP-seq中容易受到噪音干扰而无法准确检测。ChIP-DIP通过独特的抗体池设计和条形码技术,能够显著提高对这些低丰度蛋白质的检测灵敏度。
扩展应用:从单一细胞到全基因组的蛋白质-DNA相互作用分析
ChIP-DIP不仅在小规模实验中表现出色,它的可扩展性同样令人惊叹。通过改变抗体池的大小和组合,研究人员能够在同一实验中分析大量的蛋白质-DNA相互作用。例如,在K562细胞实验中,研究人员使用了52个抗体,成功捕捉到超过160种不同的蛋白质,包括转录因子、组蛋白修饰和染色质调控因子等。这种能力使得ChIP-DIP成为探索基因调控网络的强大工具。
更重要的是,ChIP-DIP技术的高灵敏度使其能够在较少的细胞样本中产生可靠的数据。在实验中,研究人员使用了从5万细胞到450万细胞的不同细胞量,结果显示,ChIP-DIP能够在仅使用5万细胞的情况下成功生成35种不同蛋白质的定位图谱。这意味着,ChIP-DIP不仅适用于常见的大规模细胞样本,也能够应用于稀有细胞类型或临床样本中的蛋白质-DNA相互作用分析。
ChIP-DIP在免疫反应中的独特应用
ChIP-DIP技术不仅适用于静态基因调控研究,还能够揭示基因调控的动态过程。在关于脂多糖(LPS)刺激小鼠骨髓来源树突状细胞(mDCs)的实验中,研究人员利用ChIP-DIP技术分析了染色质修饰在不同时间点(0小时、6小时和24小时)的动态变化。结果发现,LPS刺激后6小时,多个与基因激活相关的染色质修饰,如H3K27ac(组蛋白H3的第27位赖氨酸乙酰化)和H3K9ac(组蛋白H3的第9位赖氨酸乙酰化),发生了显著增加,尤其是在增强子区域。
更令人惊讶的是,研究还发现这些修饰的时间变化模式具有显著的不同:一些基因在6小时内快速激活并维持激活状态,而另一些基因则在24小时后才恢复原状。这一发现揭示了免疫细胞如何通过染色质的动态变化精细调控基因表达,进而响应外部刺激。
ChIP-DIP技术的出现,为基因调控的研究打开了一扇全新的大门。通过并行分析多个蛋白质-DNA相互作用,ChIP-DIP不仅显著提高了研究效率,还揭示了基因调控网络的复杂性和动态性。随着技术的不断优化和应用,ChIP-DIP有望在基因组学、转录组学、免疫学以及疾病研究等领域发挥越来越重要的作用。
参考文献
Perez, A.A., Goronzy, I.N., Blanco, M.R. et al. ChIP-DIP maps binding of hundreds of proteins to DNA simultaneously and identifies diverse gene regulatory elements. Nat Genet (2024). https://doi.org/10.1038/s41588-024-02000-5


