【论著】| 基于深度学习算法的病理学图片淋巴细胞浸润检测
时间:2024-06-09 16:00:27 热度:37.1℃ 作者:网络
[摘要] 背景与目的:深度学习方法可用来在病理切片上开展淋巴细胞的自动分割和检测。本研究探讨使用变分自编码模型预训练的方法对病理学图片进行淋巴细胞浸润检测的性能,以及去除肿瘤坏死区域对模型性能的影响。方法:使用变分自编码模型(variational auto-encoder,VAE)先在来自肿瘤基因组图谱(the Cancer Genome Atlas, TCGA)数据库中的无标注的TCGA-COAD和TCGA-READ 病理切片图像上进行预训练,获得一个自编码预训练模型,再在已标注淋巴细胞的公开数据集上进行淋巴细胞浸润检测的模型训练。为避免与坏死的区域相混淆,还训练了分割肿瘤坏死区域的Unet模型,以去除肿瘤坏死区域对淋巴细胞识别的影响。分析经过变分自编码模型预训练的淋巴细胞浸润检测模型在训练集和测试集上的受试者工作特征曲线(receiver operating characteristic curve,ROC)的曲线下面积(area under curve,AUC)及其95%置信区间(confidence interval,CI)。结果:检测模型在训练集上AUC为0.979(95% CI:0.978~0.980),准确率为92.5%(95% CI:92.3%~92.6%),Kappa值为0.849,灵敏度为0.908,特异度为0.941,精确率为0.939,召回率为0.908,F1为0.923。验证集的AUC为0.968(95% CI:0.964~0.972),准确率为91.3%(95% CI:90.6%~92.0%),Kappa值为0.826,灵敏度为0.898,特异度为0.928,精确率为0.925,召回率为0.898,F1为0.912。Resnet18模型在已标注的数据集上直接训练的结果为:验证集的准确率为83.2%(95% CI:82.2%~84.1%),Kappa值为0.664,灵敏度为0.823,特异度为0.840,精确率为0.838,召回率为0.823,F1为0.830。分割肿瘤坏死区域的Unet模型最终在训练集上的DICE值为0.78,在验证集上为0.76。去除坏死区域后本文提出的变分自编码模型预训练的淋巴细胞浸润检测模型的预测性能获得了一定的提升,在验证集上的AUC从0.968(95% CI:0.964~0.972)提升至0.971(95% CI:0.968~0.975)。准确率为92.4%(95% CI:91.7%~93.0%),Kappa值为0.849,灵敏度为0.928,特异度为0.921,精确率为0.921,召回率为0.928,F1为0.925。结论:采用变分自编码模型预训练的方法,对淋巴细胞浸润的病理学图片进行浸润检测,可获得比直接训练更优的模型表现;并且通过Unet去除肿瘤坏死区域的影响能够进一步提高模型的性能。
[关键词] 淋巴细胞浸润;人工智能;深度学习
[Abstract] Background and purpose: Deep learning methods can be used for automatic segmentation and detection of lymphocytes on pathological images. This study aimed to assess the performance of using variational autoencoding pre-training method for lymphocyte infiltration detection on pathological images, as well as the impact of removing tumor necrosis regions on model performance. Methods: Using variational autoencoding (VAE) pre-training method, pre-training was performed on a large number of unlabeled pathological images from the Cancer Genome Atlas (TCGA) database (TCGA-COAD and TCGA-READ) to obtain an auto-encoding pre-training model, and then a classifier model of lymphocyte infiltration was trained on the public data samples. To avoid confusion with necrotic regions, a Unet segmentation model for tumor necrotic regions was trained to remove the influence of tumor necrotic regions on lymphocyte identification. Results: The lymphocyte infiltration detection model pre-trained with the VAE model had an area under curve (AUC) of 0.979 (95% CI: 0.978-0.980), an accuracy of 92.5% (95% CI: 92.3%-92.6%), a kappa value of 0.849, sensitivity of 0.908, specificity of 0.941, precision of 0.939, recall of 0.908, and F1 of 0.923 under the receiver operating characteristic (ROC) curve on the training set. The AUC for the validation set was 0.968 (95% CI: 0.964-0.972), the accuracy was 91.3% (95% CI: 90.6%-92.0%), kappa value was 0.826, sensitivity was 0.898, specificity was 0.928, precision was 0.925, recall was 0.898, and F1 was 0.912. The results of Resnet18 model on the labeled dataset were as follows: accuracy of the validation set was 83.2 % (95% CI: 82.2%-84.1%), kappa value was 0.664, sensitivity was 0.823, specificity was 0.840, precision was 0.838, recall was 0.823 and F1 was 0.830. The Unet model that segmented the necrotic regions of the tumors had a final DICE of 0.78 for the training set, and 0.76 for the validation. After removing the necrotic region, the predictive performance of the pre-trained lymphocyte infiltration detection model using the VAE proposed in this article was improved to some extent, with the AUC on the validation set increasing from 0.968 (95% CI: 0.964-0.972) to 0.971 (95% CI: 0.968-0.975). The accuracy was 92.4% (95% CI: 91.7%-93.0%), kappa value was 0.849, sensitivity was 0.928, specificity was 0.921, precision was 0.921, recall was 0.928, and F1 was 0.925. Conclusion: Using the variational autoencoding model pre-training method to classify the pathological pictures of lymphocyte infiltration can obtain better model performance compared with direct training, and removing the influence of tumor necrosis areas through Unet can further improve the performance of the model.
[Key words] Lymphocyte infiltration; Artificial intelligence; Deep learning
免疫细胞和肿瘤细胞之间存在着复杂的相互作用,并且这种相互作用会影响肿瘤患者的临床疗效与预后[1]。随着肿瘤免疫治疗的迅速发展,肿瘤微环境的淋巴细胞浸润作为预后标志物变得更加重要。已有不少研究表明淋巴细胞浸润与患者预后具有相关性。在乳腺癌中,存在淋巴细胞浸润的人表皮生长因子受体2(human epidermal growth factor receptor 2,HER2)阳性和三阴性乳腺癌患者有较好的预后[2]。Meta分析[3-4]也显示,肿瘤中心或边缘的免疫细胞密度及类型与患者预后之间的差异有统计学意义。目前对淋巴细胞浸润的评估方法主要通过H-E染色或免疫组织化学检测,然后由病理学家进行计数或计算细胞密度。对淋巴细胞的浸润状态进行精准的评估需要病理学家在丰富的临床经验基础上,按照固定的流程和评估方案进行仔细评估。这在患者量少的临床试验中或许可以实施,但是在日常临床实践中,完全按照类似标准方案进行仍存在较大困难。随着计算机技术的快速发展,已有研究开始使用深度学习的方法在常规H-E染色的病理切片上开展淋巴细胞的分割和检测[5-7]。
基于深度学习方法在H-E染色的病理切片上进行淋巴细胞浸润检测主要存在两个困难,一是深度学习方法进行模型训练需一定量的标注数据。目前公开的已标注淋巴细胞的病理切片数据集数据量较少(约为86 000张图片),在训练数据较少的情况下进行模型训练容易导致过拟合;另外一个问题是淋巴细胞(lymphocyte)一般呈球形,大小不一,包括6~8 μm的小淋巴细胞、9~12 μm的中淋巴细胞和13~20 μm的大淋巴细胞,容易与肿瘤坏死的区域相混淆。
针对以上两个问题,本文使用变分自编码(variational autoencoding,VAE)模型预训练的方法,在大量无标注的病理学图像上进行预训练,获得一个自编码预训练模型,再基于公开数据样本针对淋巴细胞浸润的识别进行训练。同时为了避免与坏死的区域相混淆,本研究还训练了分割肿瘤坏死区域的Unet模型,以消除肿瘤坏死区域对淋巴细胞识别的影响。
1 材料和方法
1.1 病理切片数据来源
对于VAE模型,训练数据来自肿瘤基因组图谱(the Cancer Genome Atlas, TCGA; https://tcga-data.nci.nih.gov/)数据库中的TCGA-COAD 和 TCGA-READ 切片数据的随机截取,随机截取的切片大小为256×256像素,实际大小约为 125 μm×125 μm。
本研究中已标注淋巴细胞的病理切片数据来自公开的数据集。数据地址为(https://stonybrookmedicine.app.box.com/v/cellreportspaper)。该数据集总共包含了86 000张左右的切片,切片为500×500 像素,实际大小约为250 μm×250 μm。
1.2 输入图片预处理
模型训练时使用图像扰动进行数据增强,相关扰动如下:① 亮度扰动,亮度扰动的幅度为0.9~1.1;② 对比度扰动,对比度扰动的幅度为0.9~1.1;③ 饱和度扰动,饱和度扰动的幅度为0.9~1.1;④ 色调扰动,色调扰动的幅度为-0.1~0.1;⑤ 随机翻转,按50%的概率左右或上下翻转;⑥ 随机截取,截取的图片大小为256×256像素。数据扰动通过pytorch的torchvisions.transforms模块实现。
1.3 VAE模型架构
变分自编码模型是无监督学习模型,即无需标注数据即可进行模型训练。本研究纳入的淋巴细胞浸润标注数据约为86 000张,相对数量较少,为了获得较为稳定的模型结果,研究采用变分自编码模型先在较大的数据集上进行预训练,使得模型能够提取病理学关键信息。在预训练模型的基础上,再进行针对淋巴细胞浸润的识别模型的训练。
VAE分为编码器和解码器两部分。本研究使用的VAE的架构见图1。其中输入的图像大小为256×256像素的RGB图像,输出编码(隐向量)的数目为2 048。
编码器部分首先对切片图像进行像素重排(pixel unshuffled)。像素重排可以将图像的大小缩小到原图的1/2。减小到128×128 像素大小后,再进行一系列2D卷积(conv2d)、批次归一化(batch normalization)、Leaky激活函数(Leaky ReLu)和下采样(down sampling)操作,同时增加通道数。最后再通过两个全连接层(fully connected layer,FC),计算出图像的两个向量:一个是均值向量,一个是标准差向量。
解码器部分,首先由两组向量生成潜变量z,该潜变量的维度为2 048。该潜变量经过一个全连接层,变换结构成4×4像素的图像,再经过一系列的2D卷积(conv2d)、批次归一化 (batch normalization)、Leaky激活函数(Leaky ReLu)和上采样(up sampling),变换成128×128大小的图片,该图片包含12个通道,再通过一个pixel shuffle函数,变成3个通道256×256大小的RGB图片。
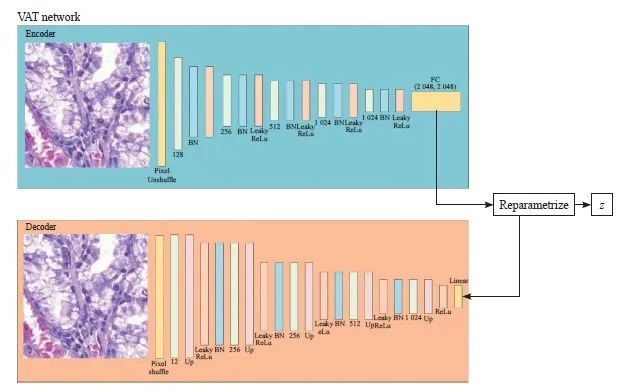
图1 VAE网络结构
Fig. 1 The network structure of VAE
1.4 VAE模型的损失函数设定
VAE训练时采用的损失函数为两个函数的组合,包括KL散度(Kullback-Leibler divergence,KL)和图像相似性的损失函数。KL散度的损失函数的公式如下:
L(θ, φ) = -KL[ qφ (z, x) || pθ(z, x) ]
其中,z是隐向量,x是输入的图像,pθ(z, x)和qφ(z, x)都是z的条件分布。
图像相似性利用平方误差(mean square error,MSE)进行计算。KL散度和结构一致性指数的比例为1e-6∶1。VAE模型更关注重建效果。
1.5 淋巴细胞浸润识别模型的训练和超参数设定
由于样本量的限制(86 000个标注数据),所以先在大样本数据集TCGA-COAD和TCGA-READ上进行VAE预训练。在得到自编码网络后,去除解码器部分,并在后端加上一个全连接层,最后输出分类结果。
训练不进行权重锁定,通过观察模型是否收敛确定模型的学习率为1e-3, 迭代次数为200次,不进行训练率的衰减,batch size为256,在Nvidia 2080 Ti 设备上进行模型训练,模型训练时间约为5 h。
因为数据集的图像数量比较大,所以我们使用95%的数据作为训练集而将剩余的5%数据作为验证集,即使用 95∶5的比例对数据集进行随机切割。95%的数据即81 700张图片为训练集,4 300张图片为验证集。
1.6 肿瘤坏死区域分割模型Unet的架构和训练
肿瘤坏死区域的识别使用分割模型进行。采用Unet对坏死区域进行分割,模型架构见图2。训练采用的批次大小(batch size)为4,初始学习率为1e-4,共训练200个epoch,不进行权重衰减。模型训练在一台搭载Intel Core i7-8700K CPU@3.7GHz以及1块GeForce GTX 1080 Ti显卡的服务器上进行。
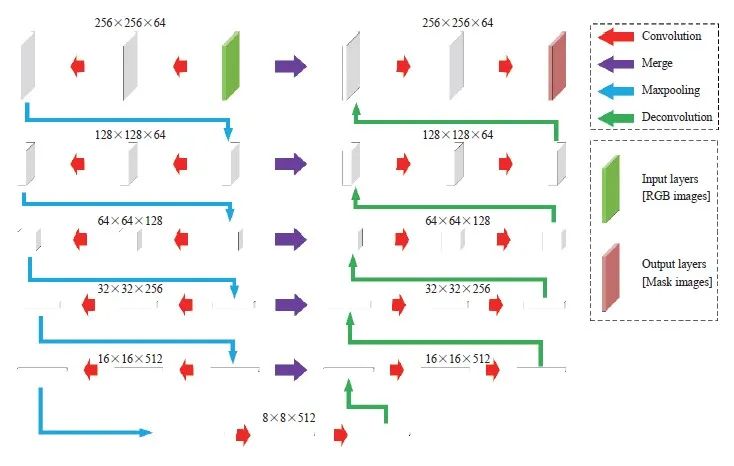
图2 Unet网络结构
Fig. 2 The network structure of Unet
1.7 淋巴细胞浸润识别模型的评估方法和指标
使用RStudio Version1.1.456对模型预测进行了全面的评估,计算了混淆矩阵(confusion matrix),预测的准确率、灵敏度、特异度、精确率、召回率和 F1,并采用受试者工作特征曲线(receiver operating characteristic curve,ROC)和曲线下面积(area under curve,AUC)呈现相关结果。
2 结 果
2.1 变分自编码模型的预训练效果
图3、图4和图5显示了验证集的1个例子,随着训练的进行,变分自编码生成的图片逐渐接近原图。

图3 原始图和1次迭代效果图
Fig. 3 The figures of original and iteration (Epoch=1)
A: 64 original images randomly captured from the validation set, with a single image size of 256×256 pixels; B: Epoch=1.

图4 50次和100次迭代效果图
Fig. 4 The figures of iterations (Epoch=50 and Epoch=100)

图5 200次和400次迭代效果图
Fig. 5 The figures of iterations (Epoch=200 and Epoch=400) A: Epoch=200; B: Epoch=400.
2.2 淋巴细胞浸润识别模型的模型训练与数值评估
分类网络训练的损失函数曲线见图6。随着训练进行,loss逐渐下降,并且准确率逐渐上升,整体没有呈现明显的过拟合现象。
最终预测模型的ROC曲线见图7。其中训练集的AUC为0.979(95% CI:0.978~0.980),验证集的AUC为0.968(95% CI:0.964~0.972)。训练集和验证集的混淆矩阵见表1:训练集的准确率为92.5%(95% CI:92.3%~92.6%),Kappa值为0.849,灵敏度为0.908,特异度为0.941,精确率为0.939,召回率为0.908,F1为0.923。验证集的准确率为91.3%(95% CI:90.6%~92.0%),Kappa值为0.826,灵敏度为0.898,特异度为0.928,精确率为0.925,召回率为0.898,F1为0.912,表2汇总了不同模型在同一数据集上的效果。
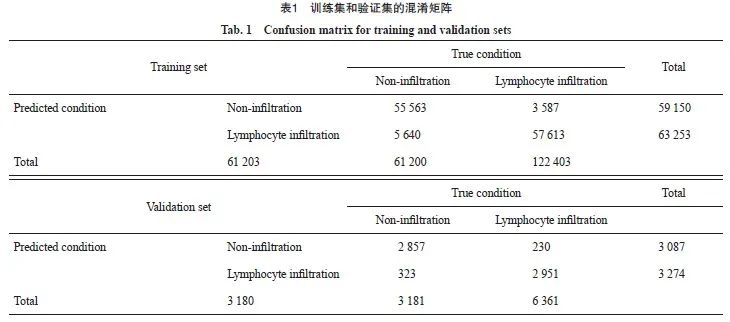
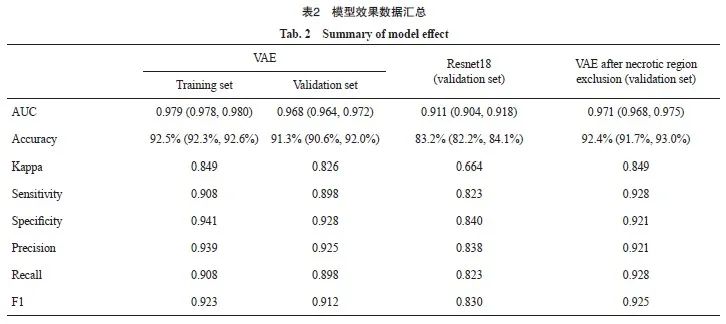
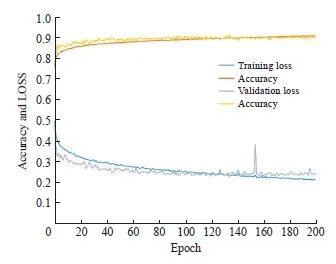
图6 分类网络训练效果
Fig.6 Effectiveness of classification network training
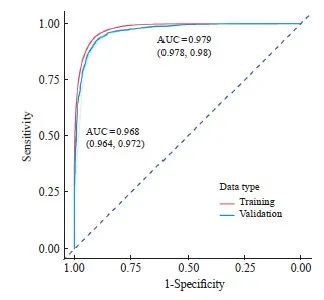
图7 最终预测模型的ROC曲线
Fig. 7 ROC curve for the final prediction model
2.3 与其他模型准确性的比较
Resnet18在相同数据集上训练的损失函数出现了明显的过拟合现象(图8)。该模型在验证集上的准确率为83.2%(95% CI:82.2%~84.1%),Kappa值为0.664,灵敏度为0.823,特异度为0.840,精确率为0.838,召回率为0.823,F1为0.830。各方面数值都要低于VAE预编码模型的训练结果。ROC曲线的比较结果见图9。
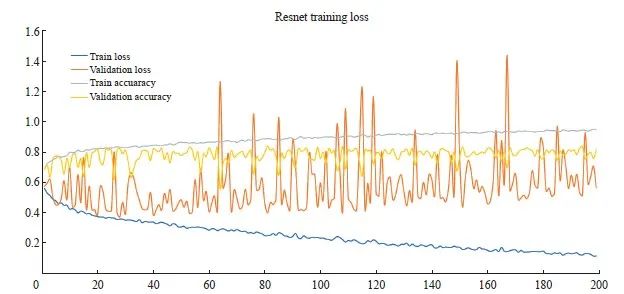
图8 Resnet训练的loss下降情况
Fig. 8 Loss drop for Resnet training
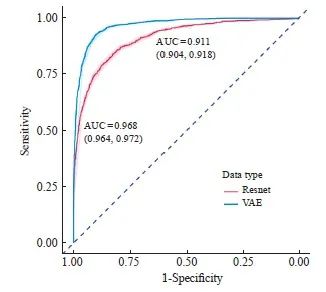
图9 VAE网络和Resnet的ROC比较
Fig. 9 ROC comparison of VAE and Resnet
2.4 肿瘤坏死区域识别模型的效果
肿瘤坏死区域识别模型的DICE值在训练集上为0.78,验证集上为0.76。图10显示了肿瘤坏死区域识别模型训练的具体效果。
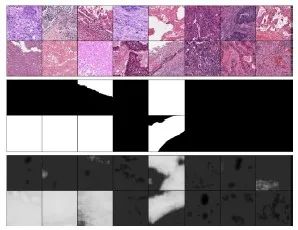
图10 验证集上肿瘤坏死区域的识别效果图
Fig. 10 The effect of identifying tumor necrotic regions in the validation set
A: The original pathology image; B: The actual tumor necrotic regions (white indicates necrotic regions and black indicates non-necrotic regions); C: The predicted necrotic regions.
2.5 肿瘤坏死区域识别对淋巴细胞浸润检测效能的提升
图11显示了去除肿瘤坏死区后进行淋巴细胞浸润检测获得的ROC曲线,去除坏死区域以后,预测效能获得提升,AUC从0.968(95% CI:0.964~0.972)提升至0.971(95% CI:0.968-0.975)。其他的数值如下,准确率为92.4%(95% CI:91.7%~93.0%),Kappa值为0.849,灵敏度为0.928,特异度为0.921,精确率为0.921,召回率为0.928,F1为0.925。
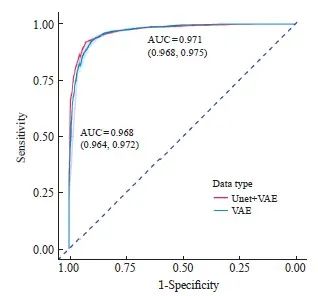
图11 去除坏死区域后进行预测获得的ROC曲线
Fig. 11 ROC curves obtained from prediction after necrotic region exclusion
3 讨 论
本研究采用变分自编码模型预训练的方法,对肿瘤病理图片进行淋巴细胞浸润检测,最终的模型准确率为92.4%。
本研究使用一个开源的数据集,相比于目前图像分类领域百万级别数据集,该数据集标注数据相对较少,而本研究采用的变分自编码预训练的方法在标注数据较少的情况下仍可取得较好的模型表现。
本研究使用自编码模型后端增加一个简单全连接层进行分类的模型架构,该模型与目前较为常见的模型架构有所差别,如Resnet、Inception、注意力架构和Transformer架构。但研究结果(表2)显示,即使采用较为简单的Resnet18模型,在模型训练阶段已显示出明显过拟合现象。因此,本研究没有对其他模型架构的表现进行测试;同时,Unet与识别网络结合部分,由于数据集标注类型的限制,只能采用两个网络分别进行处理。由于AUC以及准确率的95% CI的范围较小,同时模型截取的病理图片来自多个TCGA项目的大切片,显示模型结果较为稳定,因此,本研究没有进行交叉检验。
本研究的模型准确率较其他研究高。与本研究采用同一数据集的研究相比,Joel等[8]采用半监督的卷积神经网络方法,使用无监督的卷积自编码器进行初始化,采用一套稀疏自编码方案,将细胞核和细胞质分开处理。该自编码模型能够将输入为50 μm×50 μm(100×100像素,20倍放大率)的病理学图像编码为长度为100的向量。然后基于该网络训练淋巴细胞识别模型。该研究获得的AUC值为0.954(本研究最终AUC值为0.971)。同时该研究使用VGG直接进行训练获得的AUC值为0.923。而我们基于Resnet18进行直接训练获得AUC值为0.911,数值较为接近。进一步说明在少量的数据集上开展直接的模型训练并不能获得最优的性能。
在另一项研究中,Germán等[9]采用了较为传统的方法,认为H-E染色图像中,淋巴细胞与其他细胞最大的差别在于更小的尺寸,更圆的形状和均匀染色且染色较深。有研究[10-11]报道,利用视觉特征可能可以较好地区分淋巴细胞与其他细胞。基于染色较为均匀的特点,研究者认为细胞核图像的熵较小,并通过计算细胞形状参数面积、偏心率和长短轴比设计了需要提取的特征。基于上述人工挑选的特征,先对病理切片的颜色进行归一,再提取细胞核的特征,用支持向量机将结构分为淋巴细胞和非淋巴细胞。该方法获得的精确率为0.891,召回率为0.836,F1为0.863。研究者同时用深度学习对他们的数据集进行了测试,但该模型的表现要劣于传统的机器学习方法。值得注意的是该研究训练用的样本量偏少,只有约3 420个标注数据。
Shidan等[12]开发了一个用于进行细胞分类的软件,该软件首先识别出细胞核并将其置于画面正中,进行图像截取,识别图像中心的细胞类型,将细胞分为3类,包括肿瘤细胞、基质细胞和淋巴细胞。采用一个较为简单的深度学习模型进行训练,模型输入为80×80像素的图像。模型训练从TCGA和NLST数据集的29张全切片中提取了约11 000张图像,最后获得准确率在淋巴细胞为99.3%,基质细胞为87.9%,肿瘤细胞为91.6%。在独立数据集上获得的外部验证准确率为淋巴细胞为97.8%,基质细胞为86.5%,肿瘤细胞为85.9%。由于与本研究采用的数据集不同,无法直接比较。
本研究及其他相关研究结果显示,使用传统方法或深度学习方法都可对淋巴细胞浸润进行识别,但根据任务和训练集数据的区别,选择合适的机器学习可以获得较优的模型表现。随着数据量的增加和切片标注方法的改变,使用深度学习方法逐渐成为主流,且预测的准确率也在不断提升。同时,由于深度学习和传统方案思路技术路线有较大差异,如果将深度学习方法和传统的机器学习技术融合,模型表现仍有进一步提升的可能。
改变训练方案亦可能对模型结果存在影响,例如先训练Unet,对所有的图片进行坏死区域的识别,对坏死区域使用统一的数值进行填充,再输入分类网络进行训练,可获得更优的模型表现,相关方案可作为未来的研究方向。另一方面,虽然传统H-E染色切片中的淋巴细胞浸润评分有不需要额外染色的优势,但H-E染色无法将淋巴细胞分为细胞毒性T淋巴细胞、辅助性T淋巴细胞、调节性T淋巴细胞和其他T淋巴细胞等亚型,而T淋巴细胞亚型的区分具有一定的临床意义[13],但目前利用机器学习和人工智能对淋巴细胞亚型的识别研究较少,该方向仍需开展大量的工作。
本研究使用变分自编码模型预训练的方法,对淋巴细胞浸润的病理图片进行检测,预测模型的准确率为91.3%,AUC值为0.968;通过消除肿瘤坏死区域的影响,模型准确率提升至92.4%,AUC值为0.971。与采用Resnet18直接训练相比(模型准确率为83.2%,AUC值为0.911),本研究提出的淋巴细胞浸润识别方案准确率较高。
利益冲突声明: 所有作者均声明不存在利益冲突。
作者贡献声明:
庄晗:负责数据收集、数据分析和文章撰写;胡伟刚,章真,王佳舟:负责课题指导与论文修改。
[参考文献]
[1] FRIDMAN W H, PAGÈS F, SAUTÈS-FRIDMAN C, et al. The immune contexture in human tumours: impact on clinical outcome[J]. Nat Rev Cancer, 2012, 12(4): 298-306.
[2] SALGADO R, DENKERT C, DEMARIA S, et al. The evaluation of tumor-infiltrating lymphocytes (TILs) in breast cancer: recommendations by an International TILs Working Group 2014[J]. Ann Oncol, 2015, 26(2): 259-271.
[3] ORHAN A, VOGELSANG R P, ANDERSEN M B, et al. The prognostic value of tumour-infiltrating lymphocytes in pancreatic cancer: a systematic review and meta-analysis[J]. Eur J Cancer, 2020, 132: 71-84.
[4] FU Q F, CHEN N, GE C L, et al. Prognostic value of tumorinfiltrating lymphocytes in melanoma: a systematic review and meta-analysis[J]. Oncoimmunology, 2019, 8(7): 1593806.
[5] ALLARD M A, BACHET J B, BEAUCHET A, et al. Linear quantification of lymphoid infiltration of the tumor margin: a reproducible method, developed with colorectal cancer tissues, for assessing a highly variable prognostic factor[J]. Diagn Pathol, 2012, 7: 156.
[6] ROBINS H S, ERICSON N G, GUENTHOER J, et al. Digital genomic quantification of tumor-infiltrating lymphocytes[J]. Sci Transl Med, 2013, 5(214): 214ra169.
[7] ERIKSEN A C, ANDERSEN J B, KRISTENSSON M, et al. Computer-assisted stereology and automated image analysis for quantification of tumor infiltrating lymphocytes in colon cancer[J]. Diagn Pathol, 2017, 12(1): 65.
[8] YIN Y D, ROTHENBERG E. Probing the spatial organization of molecular complexes using triple-pair-correlation[J]. Sci Rep, 2016, 6: 30819.
[9] CORREDOR G, WANG X X, LU C, et al. A watershed and feature-based approach for automated detection of lymphocytes on lung cancer images[C]//. SPIE Medical Imaging. Proc SPIE 10581, Medical Imaging 2018: Digital Pathology, Houston, Texas, USA. 2018, 10581: 213-218.
[10] BASAVANHALLY A N, GANESAN S, AGNER S, et al. Computerized image-based detection and grading of lymphocytic infiltration in HER2+ breast cancer histopathology[J]. IEEE Trans Biomed Eng, 2010, 57(3): 642-653.
[11] KUO Y L, KO C C, LEE M J. Lymphatic infiltration detection in breast cancer H-E image prior to lymphadenectomy[J]. Biomed Eng Appl Basis Commun, 2014, 26(4): 1440007.
[12] WANG S D, WANG T, YANG L, et al. ConvPath: a software tool for lung adenocarcinoma digital pathological image analysis aided by a convolutional neural network[J]. EBioMedicine, 2019, 50: 103-110.
[13] WOUTERS M C, KOMDEUR F L, WORKEL H H, et al. Treatment regimen, surgical outcome, and T-cell differentiation influence prognostic benefit of tumor-infiltrating lymphocytes in high-grade serous ovarian cancer[J]. Clin Cancer Res, 2016, 22(3): 714-724.


